Rede da Comunidade Mundial - World Community Grid
| Desenvolvedor (s) | Krembil Research Institute |
|---|---|
| lançamento inicial | 16 de novembro de 2004 |
| Versão estável | 7.16.11 |
| Estado de Desenvolvimento | Ativo |
| Sistema operacional | Microsoft Windows , Linux , Android , macOS |
| Plataforma | BOINC |
| Modelo | Computação voluntária |
| Desempenho médio | 800 TFLOPS |
| Usuários ativos | 33.255 |
| Total de usuários | 101.949 |
| Hosts ativos | 154.119 |
| Total de hosts | 183.354 |
| Local na rede Internet | worldcommunitygrid.org |
World Community Grid ( WCG ) é um esforço para criar a maior grade de computação pública do mundo para lidar com projetos de pesquisa científica que beneficiam a humanidade. Lançado em 16 de novembro de 2004, foi coordenado pela IBM com software cliente atualmente disponível para sistemas operacionais Windows , Linux , macOS e Android . Em setembro de 2021, foi anunciado que a IBM transferiu a propriedade para o Krembil Research Institute .
O World Community Grid utiliza o poder de processamento não utilizado de dispositivos de consumo (PCs, laptops, smartphones Android, etc.) para analisar os dados criados pelos grupos de pesquisa que participam da rede. Os projetos WCG analisaram dados relacionados ao genoma humano , o microbioma humano , HIV , dengue , distrofia muscular , câncer , influenza , Ebola , Zika vírus , triagem virtual , safra de arroz , energia limpa , purificação de água e COVID-19 , entre outros áreas de pesquisa.
Existem atualmente 5 projetos ativos e 25 projetos concluídos. Vários desses projetos publicaram artigos revisados por pares com base na análise dos dados gerados pelo WCG. Isso inclui um documento do projeto OpenZika sobre a descoberta de um composto (FAM 3) que inibe a proteína NS3 Helicase do vírus Zika, reduzindo assim a replicação viral em até 86%; um artigo do FightAIDS @ home sobre a descoberta de novas vulnerabilidades na proteína HIV-1 Capsid que pode permitir um novo alvo de droga; um artigo do FightAIDS @ home sobre novas técnicas computacionais de descoberta de medicamentos para resultados mais refinados e precisos.
História
Em 2003, a IBM e outros participantes da pesquisa patrocinaram o Smallpox Research Grid Project para acelerar a descoberta de uma cura para a varíola . O estudo da varíola usou uma grade de computação distribuída maciça para analisar a eficácia dos compostos contra a varíola. O projeto permitiu aos cientistas examinar 35 milhões de moléculas de drogas potenciais contra várias proteínas da varíola para identificar bons candidatos para o desenvolvimento de tratamentos contra a varíola. Nas primeiras 72 horas, 100.000 resultados foram retornados. Ao final do projeto, 44 candidatos fortes ao tratamento foram identificados. Com base no sucesso do estudo da varíola, a IBM anunciou a criação do World Community Grid em 16 de novembro de 2004, com o objetivo de criar um ambiente técnico onde outras pesquisas humanitárias pudessem ser processadas.
O World Community Grid inicialmente suportava apenas o Windows, usando o software proprietário Grid MP da United Devices que impulsionou os projetos de computação distribuída grid.org . A demanda por suporte ao Linux levou à adição em novembro de 2005 da tecnologia de grade de código aberto Berkeley Open Infrastructure for Network Computing ( BOINC ) que alimenta projetos como SETI @ home e Climateprediction , e suporte para Mac OS X e Linux foi adicionado desde a introdução do BOINC . Em 2007, o World Community Grid migrou do Grid MP para o BOINC para todas as suas plataformas suportadas.
Em setembro de 2021, a IBM anunciou que havia transferido a propriedade do World Community Grid para o Krembil Research Institute . Todo o processo de transição ocorrerá nos próximos meses.
Dimensão do projeto
Em 21 de setembro de 2021, o World Community Grid tinha mais de 33.000 contas de usuários ativos, com mais de 154.000 dispositivos ativos. Ao longo do projeto, mais de 2.000.000 de anos cumulativos de tempo de computação foram doados e mais de 6 bilhões de unidades de trabalho foram concluídas.
Operação

O software World Community Grid usa o tempo de computação não utilizado de dispositivos conectados à Internet para realizar cálculos de pesquisa. Os usuários instalam o software cliente WCG em seus dispositivos. Este software funciona em segundo plano, usando recursos sobressalentes do sistema para processar o trabalho para WCG. Quando uma peça de trabalho ou unidade de trabalho é concluída, o software cliente o envia de volta ao WCG pela Internet e baixa uma nova unidade de trabalho. Para garantir a precisão, os servidores WCG enviam várias cópias de cada unidade de trabalho. Então, quando os resultados são recebidos, eles são coletados e validados entre si.
O World Community Grid oferece vários projetos humanitários sob um único guarda-chuva. Os usuários são incluídos em um subconjunto de projetos por padrão, mas podem optar por sair dos projetos conforme desejarem.
Quando o World Community Grid foi lançado, eles usaram o cliente Grid MP proprietário da United Devices . Depois de adicionar suporte para o cliente BOINC de código aberto em 2005, o World Community Grid acabou descontinuando o cliente Grid MP e consolidou na plataforma BOINC em 2008.
Mesmo que o WCG use software cliente de código aberto, os aplicativos reais que realizam os cálculos científicos podem não ser. No entanto, vários aplicativos científicos estão disponíveis sob uma licença gratuita, embora a fonte não esteja disponível diretamente no WCG.
Problemas potenciais
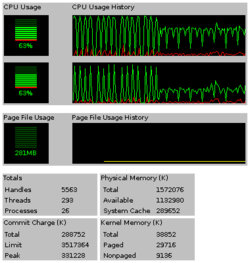
O software World Community Grid aumenta o uso da CPU , consumindo tempo de processamento não utilizado; no final dos anos 1990 e no início dos anos 2000, esses cálculos tinham como objetivo reduzir os ciclos de CPU "desperdiçados". Com CPUs modernas, onde a escala de frequência dinâmica é predominante, o uso aumentado faz com que o processador funcione em frequência mais alta, aumentando o uso de energia e o contador de aquecimento para o gerenciamento de energia . Além disso, devido a um foco crescente no desempenho de energia, ou desempenho por watt , conectar computadores antigos / ineficientes à rede aumentará a energia total / média necessária para completar os mesmos cálculos.
O cliente BOINC evita diminuir a velocidade do computador usando uma variedade de limites que suspendem a computação quando há recursos livres insuficientes. Ao contrário de outros projetos do BOINC, o World Community Grid definiu os padrões do BOINC de forma conservadora, tornando as chances de danos ao computador extremamente pequenas. A aceleração padrão da CPU é 60%. O acelerador é de granulação grossa; por exemplo, se o uso for definido como 60%, ele funcionará a 100% por 3 segundos e, a seguir, a 0% por 2 segundos, resultando em uma diminuição média do uso do processador.
Um programa complementar para computadores Windows - TThrottle - pode resolver o problema de superaquecimento, limitando diretamente o uso do computador host pelo projeto BOINC. Ele faz isso medindo a temperatura da CPU e / ou GPU e ajusta o tempo de execução de acordo. Ele também usa um tempo de comutação mais curto, de menos de um segundo, resultando em menos mudança de temperatura durante a comutação.
Estatísticas e competição
As contribuições de cada usuário são registradas e as estatísticas de contribuição do usuário são disponibilizadas publicamente. Devido ao fato de que o tempo de processamento de cada unidade de trabalho varia de computador para computador, dependendo da dificuldade da unidade de trabalho, da velocidade do computador e da quantidade de recursos ociosos disponíveis, as contribuições geralmente são medidas em termos de pontos . Os pontos são concedidos para cada unidade de trabalho, dependendo do esforço necessário para processá-la.
Ao completar uma unidade de trabalho, o cliente BOINC irá solicitar o número de pontos que ele acha que merece com base em benchmarks de software ( ver BOINC Credit System # Cobblestones ). Como vários computadores processam a mesma unidade de trabalho para garantir a precisão, os servidores do World Community Grid podem examinar os pontos reivindicados por cada um desses computadores. Os servidores WCG desconsideram os outliers estatísticos, fazem a média dos valores restantes e atribuem o número de pontos resultante a cada computador.
Na grade, os usuários podem ingressar em equipes criadas por organizações, grupos ou indivíduos. As equipes permitem um maior senso de identidade da comunidade e também podem inspirar competição. À medida que as equipes competem entre si, mais trabalho é feito para a grade em geral.
Divulgação
O World Community Grid reconhece empresas e organizações como parceiras se promoverem o WCG dentro de sua empresa ou organização. Em abril de 2021, WCG tinha 452 parceiros.
Além disso, como parte de seu compromisso com a melhoria da saúde e do bem-estar humano, os resultados de todos os cálculos concluídos no World Community Grid são liberados para o domínio público e disponibilizados para a comunidade científica.
Resultados científicos
Desde o seu lançamento, mais de trinta projetos foram executados no World Community Grid. Alguns dos resultados incluem:
- Em fevereiro de 2014, os cientistas do projeto Help Fight Childhood Cancer anunciaram a descoberta de 7 compostos que destroem as células cancerosas do neuroblastoma sem quaisquer efeitos colaterais aparentes. Esta descoberta, feita com o apoio dos voluntários WCG, é um passo positivo para um novo tratamento. O projeto anunciou que está buscando uma colaboração com uma empresa farmacêutica para desenvolver os compostos em tratamentos. Dado o sucesso do projeto, os cientistas declararam que já estão planejando um projeto de acompanhamento que se concentrará em outros cânceres pediátricos , possivelmente em colaboração com um grupo de oncologia pan-asiática recém-formado, do qual são membros fundadores.
- Em julho de 2012, o Human Proteome Folding Project publicou vários artigos usando dados do WCG. Isso inclui um artigo sobre métodos de validação e um novo banco de dados de predições de estrutura e função de proteínas; um artigo sobre a identificação de proteínas que regulam os processos humanos; um artigo sobre a análise dos genomas de cinco famílias de plantas e seus proteomas, para o qual WCG foi usado na criação de mais de 29.000 estruturas de proteínas; um artigo sobre o proteoma de Saccharomyces cerevisiae .
- O projeto GO Fight Against Malaria relatou a descoberta de várias moléculas que são eficazes contra a malária e a tuberculose resistente a medicamentos (incluindo TDR-TB, para a qual não há tratamento disponível). O projeto também testou novas moléculas contra MRSA , Filariose e Peste Bubônica . Os testes de laboratório continuam para transformar essas moléculas em possíveis tratamentos. O GFAM também foi o primeiro projeto a realizar um bilhão de cálculos de acoplamento diferentes. Um artigo foi publicado em janeiro de 2015, com mais dois pendentes de submissão. Em junho de 2015, o projeto relatou que dos dois "acertos" descobertos contra uma cepa de tuberculose resistente a medicamentos, vários "análogos" foram sintetizados, o melhor dos quais inibe o crescimento de Mycobacterium tuberculosis e é relativamente não tóxico para mamíferos células. A falta de financiamento impediu pesquisas adicionais sobre os dados.
- Os cientistas do projeto Descobrindo Drogas da Dengue - Juntos relataram a descoberta de vários novos inibidores da protease da Dengue, a maioria dos quais também inibem a protease do vírus do Nilo Ocidental . Alguns deles já entraram em "estudos pré-clínicos de farmacocinética e eficácia cruciais". Em novembro de 2014, uma atualização relatou que os cientistas têm um medicamento que desativa uma enzima chave que permite a replicação do vírus da dengue. Ele também mostrou o mesmo comportamento em outros flavivírus, como o vírus do Nilo Ocidental. Nenhum efeito colateral negativo, como toxicidade, carcinogenicidade ou mutagenicidade, foi observado, tornando este medicamento um candidato a antiviral muito forte para esses vírus. Os cientistas agora estão trabalhando para sintetizar variantes da molécula para melhorar sua atividade e entrar em testes pré-clínicos e clínicos planejados. No entanto, em uma atualização de outubro de 2018, a equipe de pesquisa relatou que nenhum de seus projetos atuais produziu um inibidor da protease da dengue altamente potente que pudesse ser testado in vivo .
- Em junho de 2013, o Projeto Energia Limpa publicou um banco de dados com mais de 2,3 milhões de moléculas orgânicas que tiveram suas propriedades caracterizadas. Destas, 35.000 moléculas mostraram potencial para dobrar a eficiência em relação às células solares orgânicas que estão sendo produzidas atualmente. Antes dessa iniciativa, os cientistas conheciam apenas um punhado de materiais à base de carbono que eram capazes de converter a luz solar em eletricidade de forma eficiente.
- Em fevereiro de 2010, os cientistas do projeto FightAIDS @ Home anunciaram que encontraram dois compostos que tornam possível uma classe potencialmente nova de drogas de combate à AIDS. Os compostos se ligam ao vírus em locais de ligação recém-descobertos e, portanto, podem ser usados para "melhorar as terapias existentes, tratar cepas da doença resistentes a medicamentos e retardar a evolução da resistência aos medicamentos no vírus."
- Em julho de 2015, o projeto Drug Search for Leishmaniose anunciou que havia testado os 10 principais compostos com maior eficiência prevista em mais de 100 identificados por meio de unidades de trabalho WCG. Destes 10, 4 mostraram "resultados positivos" em testes in vitro , com um mostrando "um resultado excepcionalmente promissor". Em agosto de 2017, os testes in vivo dos 4 compostos em hamsters mostraram resultados favoráveis, com um composto induzindo "uma cura quase completa das lesões em dois de cada cinco hamsters." No entanto, em uma atualização de março de 2018, a equipe de pesquisa anunciou que nenhum dos 10 compostos testados tinha atividade anti-leishmaniose suficiente.
- Em julho de 2015, o projeto Computing for Clean Water anunciou que um artigo havia sido publicado na revista Nature Nanotechnology descrevendo um novo tipo de filtro de água utilizando nanotubos de forma eficiente. "[Os] nanotubos são feitos de folhas de átomos de carbono com um único átomo de espessura, chamadas de grafeno , enroladas em tubos minúsculos, com diâmetros de apenas alguns nanômetros - um décimo milésimo do diâmetro de um cabelo humano. O tamanho do tubos permitem a passagem de moléculas de água, mas bloqueiam patógenos e contaminantes maiores, purificando a água. " Ao executar simulações no WCG, os cientistas descobriram que certos tipos de vibrações naturais chamados fônons , sob condições específicas, podem levar a um aumento de fluxo de água em mais de 300% através dos nanotubos, em comparação com as previsões teóricas anteriores.
- Em abril de 2015, os cientistas do projeto Say No To Schistosoma relataram que análises subsequentes foram realizadas e as três substâncias candidatas mais promissoras foram identificadas para testes in vitro .
- Em março de 2019, os pesquisadores do FightAIDS @ Home publicaram um artigo descrevendo uma "Nova interação entre subunidades crítica para HIV-1 Core Assembly" que "define um Inibidor Potencialmente Alvejado Binding Pocket". Usando o World Community Grid, mais de 1,6 milhão de compostos foram usados para atingir 20 conformações deste bolsão. Os resultados preliminares sugerem que seja um sítio de ligação plausível para compostos antivirais. Análises adicionais destes compostos são objeto de um estudo independente.
Projetos ativos
OpenPandemics - COVID-19
Em 1º de abril de 2020, a IBM anunciou o OpenPandemics - COVID-19 . O projeto visa identificar possíveis tratamentos para a síndrome respiratória aguda grave coronavírus 2 (SARS-CoV-2), responsável pela pandemia de COVID-19 . O WCG fará parceria com a Scripps Research , com quem já fez parceria no passado, principalmente em projetos do FightAIDS @ Home . O projeto é executado em CPUs e GPUs e também servirá para criar uma "ferramenta de código aberto de resposta rápida que ajudará todos os cientistas a pesquisar rapidamente por tratamentos para futuras pandemias."
O projeto foi lançado em 14 de maio de 2020.
Mapeando Marcadores de Câncer
Mapping Cancer Markers (lançado em 8 de novembro de 2013). O projeto visa identificar os marcadores associados a vários tipos de câncer e está analisando milhões de pontos de dados coletados de milhares de amostras de tecido de pacientes saudáveis e cancerosos. Estes incluem tecidos com cancros do pulmão, ovário, próstata, pâncreas e mama. Ao comparar esses diferentes pontos de dados, os pesquisadores visam identificar padrões de marcadores para diferentes tipos de câncer e correlacioná-los com diferentes resultados, incluindo capacidade de resposta a várias opções de tratamento. O projeto está se concentrando em 4 tipos de câncer, com o primeiro foco sendo o câncer de pulmão, e irá passar para o câncer de ovário, câncer de próstata e sarcoma.
FightAIDS @ Home Fase 2
FightAIDS @ Home Fase 2 (lançado em 30 de setembro de 2015) está olhando mais de perto os resultados da Fase 1. O projeto tem dois objetivos nos primeiros experimentos; a arquitetura de simulação está funcionando corretamente e fornecendo resultados confiáveis, e o uso de BEDAM e AutoDock juntos fornece melhores resultados do que usar apenas BEDAM ou AutoDock.
Ajude a parar a tuberculose
Help Stop TB foi lançado em março de 2016 para ajudar a combater a tuberculose , uma doença causada por uma bactéria que está desenvolvendo resistência aos tratamentos disponíveis atualmente. Os cálculos deste projeto têm como alvo os ácidos micólicos da capa protetora da bactéria, simulando o comportamento dessas moléculas para entender melhor como oferecem proteção à bactéria.
Esmagar o câncer infantil
Lançado em janeiro de 2017, o projeto Smash Childhood Cancer baseia-se no trabalho do projeto Help Fight Childhood Cancer, procurando por candidatos a medicamentos que visam outros cânceres infantis. Após a aposentadoria do Dr. Akira Nakagawara em março de 2020, o investigador principal mudou para o Dr. Godfrey Chan, que era um dos membros originais da equipe do Smash Childhood Cancer. Além disso, PRDM14 e Fox01 foram adicionados como novos alvos para investigação.
Projeto de chuva na África
O Africa Rainfall Project (lançado em outubro de 2019) usará o poder de computação do World Community Grid, dados da The Weather Company e outros dados para melhorar a modelagem de chuva, o que pode ajudar os agricultores na África Subsaariana a aumentar suas safras com sucesso.
Projetos completos
Enrolamento do Proteoma Humano - Fase 1
O primeiro projeto lançado no World Community Grid foi o Human Proteome Folding Project, ou HPF1, que visa prever a estrutura das proteínas humanas . O projeto foi lançado em 16 de novembro de 2004 e concluído em 18 de julho de 2006. Este projeto foi o único que a computação foi feita em conjunto com o projeto de computação distribuída grid.org . Idealizado por Richard Bonneau no Institute for Systems Biology , o projeto usou computação em grade para produzir as estruturas prováveis para cada uma das proteínas usando um Rosetta Score. A partir dessas previsões, os pesquisadores esperam prever a função de uma miríade de proteínas. Esta maior compreensão das proteínas humanas pode ser vital na busca de curas para doenças humanas . A computação para este projeto foi oficialmente concluída em 18 de julho de 2006. Os resultados da pesquisa para a porção de levedura de HPF1 foram publicados.
Enrolamento do Proteoma Humano - Fase 2
Human Proteome Folding - Fase 2 (HPF2) (lançado em 23 de junho de 2006) foi o terceiro projeto a rodar no World Community Grid, e concluído em 2013. Este projeto, na sequência do HPF1, teve como foco as proteínas secretadas por humanos , com foco especial nos biomarcadores e nas proteínas da superfície das células, bem como no Plasmodium , o organismo que causa a malária. HPF2 gera modelos de proteína de resolução mais alta do que HPF1. Embora esses modelos de resolução mais alta sejam mais úteis, eles também exigem mais poder de processamento para gerar.
Em um relatório de status de julho de 2012, os cientistas do projeto relataram que os resultados gerados pelos cálculos WCG estão sendo usados pelo Dr. Markus Landthaler do Centro Max Delbruch de Medicina Molecular (MDC) em Berlim. Os resultados do HPF2 ajudaram o Dr. Markus Landthaler e seus colaboradores a escrever um novo artigo sobre "O proteoma ligado ao mRNA e seu perfil de ocupação global em transcrições de codificação de proteínas"
Ajude a derrotar o câncer
O projeto Help Defeat Cancer visa melhorar a capacidade dos profissionais médicos de determinar as melhores opções de tratamento para pacientes com câncer de mama, cabeça ou pescoço. O projeto foi lançado em 20 de julho de 2006 e concluído em abril de 2007. O projeto funcionou identificando padrões visuais em um grande número de microarranjos de tecido retirados de amostras de tecido arquivadas. Ao correlacionar os dados do padrão com informações sobre o tratamento e o resultado do paciente, os resultados deste projeto podem ajudar a fornecer opções de tratamento mais direcionadas.
Comparação de Genoma
O projeto Comparação de Genomas é patrocinado pela instituição de pesquisa brasileira Fiocruz . O projeto foi lançado em 21 de novembro de 2006 e concluído em 21 de julho de 2007. O projeto visa comparar sequências de genes de diferentes organismos entre si, a fim de encontrar semelhanças entre eles. Os cientistas esperam descobrir para que propósito uma sequência de genes específica atende a uma função específica de um organismo, comparando-a com uma sequência de genes semelhante de função conhecida em outro organismo.
Ajuda na cura da distrofia muscular - Fase 1
Help Cure Muscular Dystrophy é administrado pela Décrypthon , uma colaboração entre a Associação Francesa de Distrofia Muscular, o Centro Nacional Francês de Pesquisa Científica e a IBM . A Fase 1 foi lançada em 19 de dezembro de 2006 e concluída em 11 de junho de 2007. O projeto investigou as interações proteína-proteína para 40.000 proteínas cujas estruturas são conhecidas, com foco particular nas proteínas que desempenham um papel nas doenças neuromusculares . O banco de dados de informações produzido ajudará os pesquisadores a projetar moléculas para inibir ou aumentar a ligação de macromoléculas específicas , esperançosamente levando a melhores tratamentos para distrofia muscular e outras doenças neuromusculares. Este projeto estava disponível apenas para agentes rodando o cliente Grid MP , tornando-o indisponível para usuários rodando BOINC .
Descobrindo Drogas Dengue - Juntos
Discovering Dengue Drugs - Together foi patrocinado por cientistas da Universidade do Texas e da Universidade de Chicago e será executado em duas fases. A Fase 1, lançada em 21 de agosto de 2007, usou AutoDock 2007 (o mesmo software usado para FightAIDS @ Home ) para testar drogas antivirais potenciais (por meio da inibição da protease NS3 ) contra vírus da família flaviviridae e concluído em 11 de agosto de 2009. Fase 2 " [usa] um programa mais intensivo em computação para selecionar os candidatos que passam pela Fase 1. " Os candidatos a medicamentos que passarem pela Fase 2 serão testados em laboratório.
AfricanClimate @ Home
A missão do AfricanClimate @ Home era desenvolver modelos climáticos mais precisos de regiões específicas da África. A intenção era servir de base para a compreensão de como o clima mudará no futuro, para que medidas destinadas a atenuar os efeitos adversos das mudanças climáticas pudessem ser implementadas. O tremendo poder de computação do World Community Grid foi usado para entender e reduzir a incerteza com a qual os processos climáticos foram simulados na África. A Fase 1 do African Climate @ Home foi lançada em 3 de setembro de 2007 e terminou em julho de 2008.
Ajude a vencer o câncer
O projeto Help Conquer Cancer (lançado em 1 de novembro de 2007) é patrocinado pelo Ontario Cancer Institute (OCI), Princess Margaret Hospital e University Health Network de Toronto, Canadá. O projeto envolve cristalografia de raios-X . A missão do Help Conquer Cancer é melhorar os resultados da cristalografia de raios-X de proteínas, que ajuda os pesquisadores não apenas a anotar partes desconhecidas do proteoma humano, mas também melhora sua compreensão da iniciação, progressão e tratamento do câncer.
O projeto HCC foi o primeiro projeto WCG a se beneficiar de unidades de processamento gráfico (GPU) s que ajudaram a terminá-lo muito mais cedo do que o inicialmente projetado devido ao enorme poder das GPUs. No relatório de status de abril de 2013, os cientistas relatam que ainda há muitos dados para analisar, mas que estão preparando um novo projeto que buscará assinaturas prognósticas e preditivas (conjuntos de genes, proteínas, microRNAs, etc.) que ajudam a prever o paciente sobrevivência e resposta ao tratamento. O projeto foi concluído em maio de 2013.
Arroz Nutritivo para o Mundo
O Arroz Nutritivo para o Mundo projeto é realizado por Ram Samudrala de Biologia Computacional Grupo de Pesquisa na Universidade de Washington . O projeto foi lançado em 12 de maio de 2008 e concluído em 6 de abril de 2010. O objetivo deste projeto é prever a estrutura das proteínas das principais variedades de arroz , a fim de ajudar os agricultores a criar melhores variedades de arroz com maior produtividade . promover maior resistência a doenças e pragas e utilizar uma gama completa de nutrientes biodisponíveis que podem beneficiar pessoas em todo o mundo, especialmente em regiões onde a desnutrição é uma preocupação crítica. O projeto foi coberto por mais de 200 meios de comunicação desde o seu início. Em 13 de abril de 2010, o World Community Grid anunciou oficialmente que o projeto Arroz Nutritivo para o Mundo terminou em 6 de abril de 2010.
Em abril de 2014, uma atualização foi postada informando que a equipe de pesquisa foi capaz de publicar informações estruturais sobre milhares de proteínas e avançar no campo da modelagem computacional de proteínas. Espera-se que esses resultados - que só foram possíveis por causa da enorme quantidade de poder computacional doado que eles tinham disponível - guiem pesquisas futuras e esforços de ciência de plantas.
O Projeto de Energia Limpa
O projeto Energia Limpa é patrocinado pelos cientistas do Departamento de Química e Biologia Química da Universidade de Harvard . A missão do Projeto Energia Limpa é encontrar novos materiais para a próxima geração de células solares e, posteriormente, dispositivos de armazenamento de energia . Os pesquisadores estão empregando mecânica molecular e cálculos de estrutura eletrônica para prever as propriedades ópticas e de transporte das moléculas que podem se tornar a próxima geração de materiais de células solares.
A Fase 1 foi lançada em 5 de dezembro de 2008 e concluída em 13 de outubro de 2009. Aproveitando o poder de computação do World Community Grid, os pesquisadores foram capazes de calcular as propriedades eletrônicas de dezenas de milhares de materiais orgânicos - muito mais do que jamais poderiam ser testado em um laboratório - e determinar quais candidatos são mais promissores para o desenvolvimento de tecnologia de energia solar acessível .
A Fase 2 foi lançada em 28 de junho de 2010, patrocinada pelos cientistas do Departamento de Química e Biologia Química da Universidade de Harvard . Outros cálculos sobre propriedades ópticas, eletrônicas e outras propriedades físicas dos materiais candidatos estão sendo conduzidos com o software de química quântica Q-Chem . Suas descobertas foram submetidas ao periódico Energy & Environmental Science .
Ajude a combater o câncer infantil
O projeto Help Fight Childhood Cancer (lançado em 13 de março de 2009) é patrocinado pelos cientistas do Chiba Cancer Center Research Institute e da Chiba University . A missão do projeto Help Fight Childhood Cancer é encontrar drogas que podem desativar três proteínas específicas associadas ao neuroblastoma , um dos tumores sólidos mais frequentes em crianças. A identificação dessas drogas pode tornar a doença muito mais curável quando combinada com o tratamento quimioterápico .
Pesquisa de drogas antivirais para influenza
O projeto Influenza Antiviral Drug Search é patrocinado pelo Dr. Stan Watowich e sua equipe de pesquisa na University of Texas Medical Branch ( Galveston , Texas , EUA). O projeto foi lançado em 5 de maio de 2009 e concluído em 22 de outubro de 2009. A missão do projeto Influenza Antiviral Drug Search é encontrar novos medicamentos que possam impedir a propagação de uma infecção por influenza no corpo. A pesquisa abordará especificamente as cepas de influenza que se tornaram resistentes aos medicamentos, bem como as novas cepas que estão surgindo. Identificar os compostos químicos que são os melhores candidatos irá acelerar os esforços para desenvolver tratamentos que seriam úteis no gerenciamento de surtos de influenza sazonal e futuras epidemias de influenza e até mesmo pandemias. A Fase 1 do projeto de Busca de Medicamentos Antivirais para Influenza já terminou em 22 de outubro de 2009. Agora os pesquisadores estão realizando o pós-processamento dos resultados da Fase 1 e se preparando para a Fase 2.
Em novembro de 2012, os cientistas do projeto afirmaram que, pelo fato de não haver perigo imediato de surto de influenza, todos os resultados do projeto seriam postados online e seus recursos seriam redirecionados para o Projeto Dengue.
Ajuda na cura da distrofia muscular - Fase 2
World Community Grid e pesquisadores apoiados pela Decrypthon, uma parceria entre AFM (Associação Francesa de Distrofia Muscular), CNRS (Centro Nacional Francês de Pesquisa Científica), Universite Pierre et Marie Curie e IBM estavam investigando interações proteína-proteína para mais de 2.200 proteínas cujos estruturas são conhecidas, com foco particular nas proteínas que desempenham um papel nas doenças neuromusculares . A Fase 2 foi lançada em 12 de maio de 2009 e concluída em 26 de setembro de 2012. O banco de dados de informações produzido ajudará os pesquisadores a projetar moléculas para inibir ou aumentar a ligação de macromoléculas específicas , esperançosamente levando a melhores tratamentos para distrofia muscular e outras doenças neuromusculares.
A fase 2 do projeto Help Cure Muscular Dystrophy começou assim que os resultados da primeira fase foram analisados. A fase 2 foi executada na plataforma BOINC .
Descobrindo as drogas da dengue - Juntos - Fase 2
Discovering Dengue Drugs - Together - Fase 2 (lançado em 17 de fevereiro de 2010) é patrocinado pela University of Texas Medical Branch (UTMB) em Galveston , Texas , Estados Unidos e pela University of Chicago em Illinois , EUA. A missão é identificar candidatos a medicamentos promissores para combater a Dengue , a Hepatite C , o Nilo Ocidental , a Febre Amarela e outros vírus relacionados. O extenso poder de computação do World Community Grid será usado para completar os cálculos de descoberta de medicamentos baseados na estrutura necessários para identificar esses candidatos a medicamentos.
Computação para Água Limpa
Computing for Clean Water (lançado em 20 de setembro de 2010) é patrocinado pelo Center for Nano e Micro Mechanics da Tsinghua University em Pequim . A missão do projeto é fornecer uma visão mais profunda da escala molecular sobre as origens do fluxo eficiente de água por meio de uma nova classe de materiais de filtro. Essa visão, por sua vez, guiará o desenvolvimento futuro de filtros de água de baixo custo e mais eficientes. Estima-se que 1,2 bilhão de pessoas não tenham acesso a água potável e 2,6 bilhões tenham pouco ou nenhum saneamento. Como resultado, milhões de pessoas morrem anualmente - cerca de 3.900 crianças por dia devido à falta de água potável. Em 25 de abril de 2014, os cientistas do projeto divulgaram uma atualização informando que eles tinham resultados empolgantes para relatar quando o artigo fosse enviado e que o projeto no WCG estava concluído.
Pesquisa de drogas para leishmaniose
O Drug Search for Leishmaniosis (lançado em 7 de setembro de 2011) é liderado pela Universidade de Antioquia em Medellín , Colômbia , com a assistência de pesquisadores da University of Texas Medical Branch em Galveston, Texas. A missão é identificar potenciais candidatos a moléculas que possam ser desenvolvidos em tratamentos para leishmaniose . O extenso poder de computação do World Community Grid será usado para realizar simulações de computador das interações entre milhões de compostos químicos e certas proteínas-alvo. Isso ajudará a encontrar os compostos mais promissores que podem levar a tratamentos eficazes para a doença.
Projeto GO de luta contra a malária
A missão do projeto GO Fight Against Malaria (lançado em 16 de novembro de 2011) é descobrir candidatos a medicamentos promissores que possam ser desenvolvidos em novos medicamentos que curem formas resistentes de malária . O poder de computação do World Community Grid será usado para realizar simulações de computador das interações entre milhões de compostos químicos e certas proteínas-alvo, para prever sua capacidade de eliminar a malária. Os melhores compostos serão testados por cientistas do The Scripps Research Institute de La Jolla, Califórnia, EUA e posteriormente desenvolvidos em possíveis tratamentos para a doença.
Diga não ao Schistosoma
Diga Não ao Schistosoma (lançado em 22 de fevereiro de 2012) foi o 20º projeto de pesquisa a ser lançado no World Community Grid. Os pesquisadores da Universidade Infórium em Belo Horizonte e FIOCRUZ-Minas , Brasil , correu este projecto no World Community Grid para realizar simulações de computador das interacções entre milhões de compostos químicos e de certas proteínas-alvo na esperança de encontrar tratamentos eficazes para esquistossomose . Em abril de 2015, análises subsequentes foram realizadas e as três substâncias candidatas mais promissoras foram identificadas para testes in vitro.
Computação para Água Sustentável
Computing for Sustainable Water foi o 21º projeto de pesquisa a ser lançado no World Community Grid. Os pesquisadores da University of Virginia estavam executando este projeto no World Community Grid para estudar os efeitos da atividade humana em uma grande bacia hidrográfica e obter percepções mais profundas sobre quais ações podem apoiar a restauração, saúde e sustentabilidade deste importante recurso hídrico. O projeto foi lançado em 17 de abril de 2012 e concluído em 17 de outubro de 2012.
Desvendando Mistérios do Genoma
O projeto Uncovering Genome Mysteries foi lançado em 16 de outubro de 2014 e é uma colaboração conjunta entre cientistas brasileiros e australianos. O projeto visa examinar cerca de 200 milhões de genes de várias formas de vida e compará-los com genes conhecidos para descobrir qual é sua função. Os resultados podem ter efeito em campos como a medicina e a pesquisa ambiental.
Superar o ebola juntos
Outsmart Ebola Together foi uma colaboração com o Scripps Research Institute para ajudar a encontrar compostos químicos para combater o vírus Ebola . Foi lançado em 3 de dezembro de 2014. O objetivo é bloquear etapas cruciais no ciclo de vida do vírus, encontrando drogas com alta afinidade de ligação com algumas de suas proteínas. Existem dois alvos: uma proteína de superfície usada pelo vírus para infectar células humanas e proteínas "transformadoras" que mudam de forma para realizar funções diferentes. O projeto foi concluído oficialmente em 6 de dezembro de 2018.
OpenZika
O OpenZika foi lançado em 18 de maio de 2016 para ajudar a combater o vírus Zika . O projeto visa proteínas que se acredita serem usadas pelo vírus Zika para sobreviver e se espalhar no corpo, com base em resultados conhecidos de doenças semelhantes, como dengue e febre amarela . Esses resultados ajudarão os pesquisadores a desenvolver um medicamento anti-zika. O projeto foi concluído oficialmente em 13 de dezembro de 2019.
FightAIDS @ Home
FightAIDS @ Home (lançado em 19 de novembro de 2005) foi o segundo projeto do World Community Grid e o primeiro a ter como alvo uma única doença. Cada computador individual processa uma molécula de droga potencial e testa quão bem ela se encaixaria na protease do HIV , agindo como um inibidor da protease . O Scripps Research Institute publicou seu primeiro artigo científico revisado por pares sobre os resultados do FightAIDS @ Home em 21 de abril de 2007. Este artigo explica que os resultados até aquele ponto serão usados principalmente para melhorar a eficiência dos cálculos futuros do FightAIDS @ Home.
Projeto de imunidade a microbiota
Microbiome Immunity Project (lançado em agosto de 2017) é um estudo de proteínas em bactérias localizadas no corpo humano; o microbioma humano , que é feito de cerca de 3 milhões de genes bacterianos separados. Ao aprender os genes das bactérias, suas formas individuais podem ser conhecidas, e cada forma física determina a função das bactérias. As instituições colaborativas incluem a Universidade da Califórnia em San Diego , o Broad Institute of MIT e Harvard e o Flatiron Institute da Simons Foundation .
Veja também
- BOINC
- Folding @ home
- Questão global
- Lista de projetos de computação distribuída
- Computação voluntária
- Comunidade mundial
