Fosforilação de proteínas - Protein phosphorylation
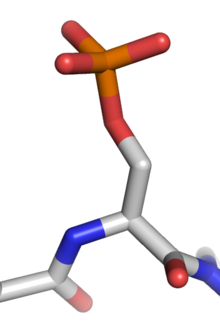
A fosforilação de proteínas é uma modificação reversível pós-tradução de proteínas em que um resíduo de aminoácido é fosforilado por uma proteína quinase pela adição de um grupo fosfato ligado covalentemente. A fosforilação altera a conformação estrutural de uma proteína, fazendo com que ela seja ativada, desativada ou modificando sua função. Aproximadamente 13.000 proteínas humanas têm locais que são fosforilados.
A reação reversa de fosforilação é chamada de desfosforilação e é catalisada por proteínas fosfatases . As proteínas quinases e fosfatases atuam independentemente e em equilíbrio para regular a função das proteínas.
Os aminoácidos mais comumente fosforilados são serina , treonina , tirosina em eucariotos e também histidina em procariotos e plantas (embora agora seja conhecido por ser comum em humanos). Essas fosforilações desempenham papéis importantes e bem caracterizados nas vias de sinalização e no metabolismo. No entanto, outros amino ácidos também podem ser fosforilados após a tradução, incluindo a arginina , lisina , ácido aspártico , ácido glutâmico e cisteína , e estes fosforilada amino ácidos foram recentemente identificados para estar presente em extractos de células humanas e fixos células humanas, utilizando uma combinação de análise baseada em anticorpos (para pHis) e espectrometria de massa (para todos os outros aminoácidos).
A fosforilação de proteínas foi relatada pela primeira vez em 1906 por Phoebus Levene no Rockefeller Institute for Medical Research com a descoberta da vitelina fosforilada . No entanto, passaram quase 50 anos até que a fosforilação enzimática de proteínas por proteínas quinases fosse descoberta.
História
Em 1906, Phoebus Levene , do Rockefeller Institute for Medical Research, identificou o fosfato na proteína vitelina (fosvitina) e, em 1933, detectou a fosfoserina na caseína , com Fritz Lipmann. No entanto, demorou mais 20 anos até que Eugene P. Kennedy descreveu a primeira 'fosforilação enzimática de proteínas'. A primeira enzima fosforilase foi descoberta por Carl e Gerty Cori no final dos anos 1930. Carl e Gerty Cori encontraram duas formas de glicogênio fosforilase que chamaram de A e B, mas não entenderam corretamente o mecanismo da conversão da forma B em A. A interconversão da fosforilase b em fosforilase a foi posteriormente descrita por Edmond Fischer e Edwin Krebs , bem como, Wosilait e Sutherland , envolvendo um mecanismo de fosforilação / desfosforilação. Verificou-se que uma enzima, denominada fosforilase quinase e Mg-ATP, era necessária para fosforilar a glicogênio fosforilase auxiliando na transferência do grupo γ-fosforil do ATP para um resíduo de serina na fosforilase b. A proteína fosfatase 1 é capaz de catalisar a desfosforilação de enzimas fosforiladas removendo o grupo fosfato. Earl Sutherland explicou em 1950 que a atividade da fosforilase foi aumentada e, portanto, a glicogenólise estimulada quando fatias de fígado foram incubadas com adrenalina e glucagon. A fosforilação foi considerada um mecanismo de controle específico para uma via metabólica até a década de 1970, quando Lester Reed descobriu que o complexo piruvato desidrogenase mitocondrial era inativado pela fosforilação. Também na década de 1970, o termo fosforilação multisite foi cunhado em resposta à descoberta de proteínas que são fosforiladas em dois ou mais resíduos por duas ou mais quinases. Em 1975, foi demonstrado que proteínas quinases dependentes de cAMP fosforilam resíduos de serina em motivos de sequência de aminoácidos específicos. Ray Erikson descobriu que v-Src era uma quinase e Tony Hunter descobriu que v-Src fosforilava resíduos de tirosina em proteínas na década de 1970. No início de 1980, a sequência de aminoácidos da primeira proteína quinase foi determinada, o que ajudou os geneticistas a entender as funções dos genes reguladores. No final da década de 1980 e início da década de 1990, a primeira proteína tirosina fosfatase (PTP1B) foi purificada e a descoberta, bem como a clonagem de JAK quinases, foi realizada, o que levou muitos na comunidade científica a nomear a década de 1990 como a década da proteína quinase cascatas. Edmond Fischer e Edwin Krebs receberam o prêmio Nobel em 1992 “por suas descobertas sobre a fosforilação reversível de proteínas como um mecanismo regulador biológico”.
Abundância
A fosforilação reversível de proteínas é abundante em organismos procarióticos e ainda mais em organismos eucarióticos . Por exemplo, em bactérias, acredita-se que 5 a 10% de todas as proteínas sejam fosforiladas. Em contraste, estima-se que um terço de todas as proteínas humanas é fosforilado em qualquer ponto do tempo, com 230.000, 156.000 e 40.000 locais de fosforilação únicos existentes em humanos, camundongos e leveduras, respectivamente. Na levedura, cerca de 120 quinases (de aproximadamente 6.000 proteínas no total) causam 8.814 eventos de fosforilação regulados conhecidos, gerando cerca de 3.600 fosfoproteínas (cerca de 60% de todas as proteínas de levedura). Portanto, a fosforilação é um mecanismo regulatório universal que afeta uma grande parte das proteínas. Mesmo que uma proteína não seja fosforilada, suas interações com outras proteínas podem ser reguladas pela fosforilação dessas proteínas em interação.
Mecanismos e funções de fosforilação
A fosforilação introduz um grupo carregado e hidrofílico na cadeia lateral de aminoácidos, possivelmente mudando a estrutura de uma proteína ao alterar as interações com os aminoácidos próximos. Algumas proteínas, como a p53, contêm vários locais de fosforilação, facilitando a regulação complexa de vários níveis. Devido à facilidade com que as proteínas podem ser fosforiladas e desfosforiladas, esse tipo de modificação é um mecanismo flexível para as células responderem a sinais externos e condições ambientais.
As cinases fosforilam proteínas e as fosfatases desfosforilam as proteínas. Muitas enzimas e receptores são "ligados" ou "desligados" por fosforilação e desfosforilação. A fosforilação reversível resulta em uma mudança conformacional na estrutura de muitas enzimas e receptores , fazendo com que sejam ativados ou desativados. A fosforilação geralmente ocorre em resíduos de serina , treonina , tirosina e histidina em proteínas eucarióticas. A fosforilação de histidina de proteínas eucarióticas parece ser muito mais frequente do que a fosforilação de tirosina. Nas proteínas procarióticas, a fosforilação ocorre nos resíduos de serina, treonina, tirosina, histidina ou arginina ou lisina . A adição de uma molécula de fosfato (PO 4 3- ) a um grupo R apolar de um resíduo de aminoácido pode transformar uma porção hidrofóbica de uma proteína em uma porção polar e extremamente hidrofílica de uma molécula. Desta forma, a dinâmica da proteína pode induzir uma mudança conformacional na estrutura da proteína por meio de alosteria de longo alcance com outros resíduos hidrofóbicos e hidrofílicos na proteína.
Um exemplo do papel regulador que a fosforilação desempenha é a proteína supressora de tumor p53 . A proteína p53 é fortemente regulada e contém mais de 18 locais de fosforilação diferentes. A ativação do p53 pode levar à interrupção do ciclo celular, que pode ser revertida em algumas circunstâncias, ou morte celular por apoptose. Essa atividade ocorre apenas em situações em que a célula está danificada ou a fisiologia está alterada em indivíduos normais e saudáveis.
Ao sinal de desativação, a proteína torna-se desfosforilada novamente e para de funcionar. Este é o mecanismo em muitas formas de transdução de sinal , por exemplo, a maneira pela qual a luz que entra é processada nas células sensíveis à luz da retina .
As funções regulatórias da fosforilação incluem:
-
Termodinâmica biológica de reações que requerem energia
- Fosforilação de Na + / K + -ATPase durante o transporte de íons sódio (Na + ) e potássio (K + ) através da membrana celular em osmorregulação para manter a homeostase do conteúdo de água do corpo.
- Medeia a inibição enzimática
- Fosforilação da enzima GSK-3 por AKT (proteína quinase B) como parte da via de sinalização da insulina.
- A fosforilação da tirosina quinase src (pronuncia-se "sarc") pela Src quinase C-terminal (Csk) induz uma mudança conformacional na enzima, resultando em uma dobra na estrutura, que mascara seu domínio quinase e é, portanto, "desligada".
Transporte de membrana
- Fosforilação de Na + / K + -ATPase durante o transporte de íons sódio (Na + ) e potássio (K + ) através da membrana celular em osmorregulação para manter a homeostase do conteúdo de água do corpo.
- Transportador de cassete de ligação de ATP
Degradação de proteínas
- A fosforilação da arginina pela quinase McsB marca as proteínas para degradação por uma protease Clp . O sistema de fosforilação da arginina, que é amplamente distribuído pelas bactérias Gram-positivas , parece ser funcionalmente análogo ao sistema ubiquitina-proteassoma eucariótico .
Regulação enzimática (ativação e inibição)
- O primeiro exemplo de regulação de proteínas por fosforilação a ser descoberto foi a glicogênio fosforilase . Eddie Fisher e Ed Krebs descreveram como a fosforilação do glicogênio fosforilase b converteu-o no glicogênio fosforilase a ativo. Logo foi descoberto que a glicogênio sintase, outra enzima metabólica, é inativada por fosforilação.
- Fosforilação da enzima GSK-3 por AKT (proteína quinase B) como parte da via de sinalização da insulina.
- A fosforilação da tirosina quinase Src (pronuncia-se "sarc") por Csk (Src quinase C-terminal) inativa Src induzindo uma mudança conformacional que mascara seu domínio quinase.
- A fosforilação das histonas H2AX na serina 139, dentro de dois milhões de bases (0,03% da cromatina) em torno de uma quebra de fita dupla no DNA, é necessária para o reparo da quebra de fita dupla. A fosforilação de metilpurina DNA glicosilase na serina 172 é necessária para o reparo de excisão de base de dano de base alquilada.
Interações proteína-proteína
- A fosforilação dos componentes citosólicos da NADPH oxidase , uma grande enzima multiproteica ligada à membrana, presente nas células fagocíticas , desempenha um papel importante na regulação das interações proteína-proteína na enzima.
- Importante na degradação de proteínas.
- No final da década de 1990, foi reconhecido que a fosforilação de algumas proteínas faz com que elas sejam degradadas pela via ubiquitina / proteassoma dependente de ATP . Estas proteínas alvo tornam-se substratos para ligases de ubiquitina E3 particulares apenas quando são fosforiladas.
Redes de sinalização
Elucidar eventos de fosforilação de vias de sinalização complexas pode ser difícil. Nas vias de sinalização celular , a proteína A fosforila a proteína B e B fosforila C. No entanto, em outra via de sinalização, a proteína D fosforila A ou fosforila a proteína C. Abordagens globais, como a fosfoproteômica , o estudo de proteínas fosforiladas, que é um sub- ramificação da proteômica , combinada com a proteômica baseada em espectrometria de massa , tem sido utilizada para identificar e quantificar mudanças dinâmicas em proteínas fosforiladas ao longo do tempo. Essas técnicas estão se tornando cada vez mais importantes para a análise sistemática de redes de fosforilação complexas. Eles foram usados com sucesso para identificar mudanças dinâmicas no estado de fosforilação de mais de 6.000 locais após estimulação com fator de crescimento epidérmico . Outra abordagem para compreender a Rede de Fosforilação é medir as interações genéticas entre várias proteínas fosforilantes e seus alvos. Isso revela padrões recorrentes interessantes de interações - motivos de rede. Métodos computacionais foram desenvolvidos para modelar redes de fosforilação e prever suas respostas sob diferentes perturbações.
Fosforilação de histonas
O DNA eucariótico é organizado com proteínas histonas em complexos específicos chamados cromatina. A estrutura da cromatina funciona e facilita o empacotamento, organização e distribuição do DNA eucariótico. No entanto, tem um impacto negativo em vários processos biológicos fundamentais, como transcrição, replicação e reparo de DNA, restringindo a acessibilidade de certas enzimas e proteínas. A modificação pós-tradução das histonas, como a fosforilação das histonas, demonstrou modificar a estrutura da cromatina ao alterar as interações proteína: DNA ou proteína: proteína. As modificações pós-tradução das histonas modificam a estrutura da cromatina. A fosforilação da histona mais comumente associada ocorre durante as respostas celulares ao dano ao DNA, quando a histona H2A fosforilada separa grandes domínios da cromatina ao redor do local da quebra do DNA. Os pesquisadores investigaram se as modificações das histonas impactam diretamente a transcrição direcionada da RNA polimerase II. Os pesquisadores escolheram proteínas que são conhecidas por modificar as histonas para testar seus efeitos na transcrição e descobriram que a quinase induzida por estresse, MSK1, inibe a síntese de RNA. A inibição da transcrição por MSK1 foi mais sensível quando o modelo estava na cromatina, uma vez que os modelos de DNA que não estavam na cromatina eram resistentes aos efeitos do MSK1. Foi demonstrado que MSK1 fosforilou a histona H2A na serina 1 e a mutação da serina 1 em alanina bloqueou a inibição da transcrição por MSK1. Assim, os resultados sugeriram que a acetilação das histonas pode estimular a transcrição pela supressão de uma fosforilação inibitória por uma quinase como MSK1.
Quinases
Dentro de uma proteína, a fosforilação pode ocorrer em vários aminoácidos . A fosforilação em serina é considerada a mais comum, seguida pela treonina . A fosforilação da tirosina é relativamente rara, mas está no topo de muitas vias de sinalização da fosforilação de proteínas (por exemplo, em receptores ligados à tirosina quinase) na maioria dos eucariotos. A fosforilação em aminoácidos, como serina, treonina e tirosina resulta na formação de uma fosfoproteína, quando o grupo fosfato da fosfoproteína reage com o grupo -OH de uma cadeia lateral de Ser, Thr ou Tyr em uma reação de esterificação . No entanto, uma vez que as proteínas fosforiladas em tirosina são relativamente fáceis de purificar usando anticorpos , os locais de fosforilação em tirosina são relativamente bem compreendidos. A fosforilação de histidina e aspartato ocorre em procariotos como parte da sinalização de dois componentes e, em alguns casos, em eucariotos em algumas vias de transdução de sinal. A análise de histidina fosforilada usando abordagens bioquímicas e espectrométricas de massa padrão é muito mais desafiadora do que a de Ser, Thr ou Tyr. e Em procariotos, arquea e alguns eucariotos inferiores, o nitrogênio da histidina age como um nucleófilo e se liga a um grupo fosfato. Uma vez que a histidina é fosforilada, o domínio regulador do regulador de resposta catalisa a transferência do fosfato para o aspartato.
Receptor de tirosina quinases
Embora a fosforilação da tirosina seja encontrada em abundância relativamente baixa, ela é bem estudada devido à facilidade de purificação da fosfotirosina usando anticorpos . Os receptores tirosina quinases são uma importante família de receptores de superfície celular envolvidos na transdução de sinais extracelulares, como hormônios, fatores de crescimento e citocinas. A ligação de um ligante a um receptor monomérico tirosina quinase estabiliza as interações entre dois monômeros para formar um dímero , após o qual os dois receptores ligados fosforilam resíduos de tirosina em trans . A fosforilação e ativação do receptor ativam uma via de sinalização por meio da atividade enzimática e das interações com proteínas adaptadoras. A sinalização por meio do receptor do fator de crescimento epidérmico (EGFR) , um receptor tirosina quinase, é crítica para o desenvolvimento de múltiplos sistemas orgânicos, incluindo pele, pulmão, coração e cérebro. A sinalização excessiva através da via EGFR é encontrada em muitos cânceres humanos.
Quinases dependentes de ciclina
As quinases dependentes de ciclina (CDKs) são quinases serina-treonina que regulam a progressão através do ciclo celular eucariótico . Os CDKs são cataliticamente ativos apenas quando ligados a uma ciclina reguladora . As células animais contêm pelo menos nove CDKs distintos que se ligam a várias ciclinas com especificidade considerável. Os inibidores de CDK (CKIs) bloqueiam a atividade da quinase no complexo ciclina-CDK para interromper o ciclo celular em G1 ou em resposta a sinais ambientais ou danos ao DNA. A atividade de diferentes CDKs ativa as vias de sinalização celular e fatores de transcrição que regulam eventos-chave na mitose, como a transição de fase G1 / S. Os complexos de ciclina-CDK anteriores fornecem o sinal para ativar os complexos de ciclina-CDK subsequentes.
Sites
Existem milhares de locais de fosforilação distintos em uma determinada célula desde:
- Existem milhares de tipos diferentes de proteínas em qualquer célula específica (como um linfócito ).
- Estima-se que 1/10 a 1/2 das proteínas sejam fosforiladas (em algum estado celular).
- Estudos independentes indicam que 30-65% das proteínas no genoma humano e ~ 50% das proteínas no genoma da levedura podem ser fosforiladas.
- Uma análise estatística de vários experimentos de alto e baixo rendimento estima que 230.000 156.000 e 40.000 locais de fosforilação existem em humanos, camundongos e leveduras, respectivamente
- A fosforilação geralmente ocorre em vários locais distintos em uma determinada proteína.
Uma vez que a fosforilação de qualquer local em uma determinada proteína pode alterar a função ou localização dessa proteína, compreender o "estado" de uma célula requer o conhecimento do estado de fosforilação de suas proteínas. Por exemplo, se o aminoácido Serina-473 ("S473") na proteína AKT for fosforilado, AKT é, em geral, funcionalmente ativo como uma quinase. Caso contrário, é uma quinase inativa.
Os locais de fosforilação são cruciais para as proteínas e seu transporte e funções. Eles são a modificação covalente de proteínas por meio de fosforilação reversível. Isso permite que as proteínas permaneçam inbound dentro de uma célula, uma vez que o local fosforilado negativo impede sua permeabilidade através da membrana celular. A desfosforilação de proteínas permite que a célula reponha os fosfatos por meio da liberação de pirofosfatos, o que economiza o uso de ATP na célula. Um exemplo de enzima fosforiladora é encontrado na bactéria E. coli . Possui fosfatase alcalina na região periplasmática de sua membrana. A membrana mais externa é permeável a moléculas fosforiladas, porém a membrana citoplasmática interna é impermeável devido a grandes cargas negativas. Desta forma, a bactéria E. coli armazena proteínas e pirofosfatos em sua membrana periplasmática até que sejam necessários dentro da célula.
Avanços recentes na identificação fosfoproteômica resultaram na descoberta de inúmeros locais de fosforilação em proteínas. Isso exigiu um meio integrativo para dados acessíveis em que sítios de fosforilação conhecidos de proteínas são organizados. Foi criada uma base de dados com curadoria de dbPAF, contendo locais de fosforilação conhecidos em H. sapiens , M. musculus , R. norvegicus , D. melanogaster , C. elegans , S. pombe e S. cerevisiae . O banco de dados atualmente contém 294.370 locais de fosforilação não redundantes de 40.432 proteínas. Outras ferramentas de previsão de fosforilação em proteínas incluem NetPhos para eucariotos , NetPhosBac para bactérias e ViralPhos para vírus.
Serina / Treonina
Existe uma grande variedade de resíduos de serina, e a fosforilação de cada resíduo pode levar a diferentes consequências metabólicas.
- A proteína quinase N1 é responsável pela fosforilação do fator associado ao receptor TNF (TRAF1) na serina 139 sob condições específicas. O TRAF1 murino também é fosforilado pela mesma quinase, o que leva ao silenciamento da atividade de IKK / NF-κB. A eliminação da fosforilação na serina 139 pode ser alcançada pela substituição de TRAF1 por um resíduo de Alanina, o que consequentemente leva ao recrutamento melhorado de TBK1.
- No resíduo de serina 789, o FGFR1 é fosforilado por RSK2 quando a quinase está em sua forma ativa. As capacidades de sinalização do FGFR1 no local da serina 777 podem ser enfraquecidas pela fosforilação. A serina 1047 e a serina 1048 foram associadas à diminuição da afinidade de ligação da ubiquitina ligase c-Cbl a EFGR quando são fosforiladas.
- Quando a serina 349 é fosforilada, a afinidade de ligação entre o complexo de proteínas p62 e a proteína Keap1 é reforçada, o que está ligado à resposta ao estresse.
- Quando a serina 337 é fosforilada pela proteína quinase A in vitro, a eficiência de ligação ao DNA da subunidade p50 de NF-κB é grandemente aumentada.
A fosforilação de resíduos de serina e treonina é conhecida por crosstalk com modificação O- GlcNAc de resíduos de serina e treonina.
Tirosina
A fosforilação da tirosina reage rapidamente e a reação pode ser revertida. Sendo um dos principais mecanismos reguladores na transdução de sinal - crescimento celular , diferenciação , migração e homeostase metabólica são processos celulares mantidos pela fosforilação da tirosina. A função das proteínas tirosina quinases e da proteína tirosina fosfatase contrabalança o nível de fosfotirosina em qualquer proteína. O mau funcionamento de cadeias específicas de proteína tirosina quinases e proteína tirosina fosfatase tem sido associado a várias doenças humanas, como obesidade , resistência à insulina e diabetes mellitus tipo 2 . A fosforilação da tirosina não ocorre apenas em eucariotos, mas descobriu-se que ocorre em uma seleção de espécies bacterianas e está presente entre os procariotos . A fosforilação da tirosina mantém a regulação celular nas bactérias semelhante à sua função nos eucariotos.
Arginina
A fosforilação da arginina em muitas bactérias Gram-positivas marca proteínas para degradação por uma protease Clp .
Fosforilação não canônica em His, Asp, Cys, Glu, Arg e Lys em células humanas
Estudos recentes do laboratório Claire E Eyers confirmam a fosforilação generalizada de proteínas humanas em vários aminoácidos não canônicos, incluindo motivos contendo histidina fosforilada (posições 1 e 3), aspartato, cisteína, glutamato, arginina e lisina em extratos de células HeLa. Devido à instabilidade química e térmica desses resíduos fosforilados, procedimentos especiais e técnicas de separação são necessários para preservação juntamente com a fosforilação Ser, Thr e Tyr 'clássica' estável ao calor.
Detecção e caracterização
Os anticorpos podem ser usados como uma ferramenta poderosa para detectar se uma proteína é fosforilada em um determinado local. Os anticorpos ligam-se e detectam alterações conformacionais induzidas por fosforilação na proteína. Esses anticorpos são chamados de anticorpos fosfo-específicos; centenas desses anticorpos já estão disponíveis. Eles estão se tornando reagentes críticos tanto para pesquisas básicas quanto para diagnósticos clínicos.
Isoformas de modificação pós-tradução (PTM) são facilmente detectadas em géis 2D . De fato, a fosforilação substitui grupos hidroxila neutros em serinas, treoninas ou tirosinas por fosfatos carregados negativamente com pKs próximos a 1,2 e 6,5. Assim, abaixo de pH 5,5, os fosfatos adicionam uma única carga negativa; perto do pH 6,5, eles adicionam 1,5 cargas negativas; acima de pH 7,5, eles adicionam 2 cargas negativas. A quantidade relativa de cada isoforma também pode ser fácil e rapidamente determinada a partir da intensidade da coloração em géis 2D.
Em alguns casos muito específicos, a detecção da fosforilação como uma mudança na mobilidade eletroforética da proteína é possível em géis SDS-PAGE unidimensionais simples, como é descrito, por exemplo, para um coativador transcricional por Kovacs et al. Acredita-se que fortes mudanças conformacionais relacionadas à fosforilação (que persistem em soluções contendo detergentes) sejam a base desse fenômeno. A maioria dos locais de fosforilação para os quais tal mudança de mobilidade foi descrita cai na categoria de locais SP e TP (isto é, um resíduo de prolina segue o resíduo de serina ou treonina fosforilado).
Mais recentemente, análises de espectrometria de massa em grande escala foram usadas para determinar locais de fosforilação de proteínas. Nos últimos 4 anos, dezenas de estudos foram publicados, cada um identificando milhares de sites, muitos dos quais anteriormente não descritos. A espectrometria de massa é ideal para tais análises usando fragmentação HCD ou ETD , pois a adição de fosforilação resulta em um aumento na massa da proteína e do resíduo fosforilado. Espectrômetros de massa avançados e altamente precisos são necessários para esses estudos, limitando a tecnologia a laboratórios com espectrômetros de massa de última geração. No entanto, a análise de peptídeos fosforilados por espectrometria de massa ainda não é tão simples quanto para peptídeos "regulares" não modificados. Recentemente, o EThcD foi desenvolvido combinando transferência de elétrons e dissociação de colisão de alta energia. Em comparação com os métodos de fragmentação usuais, o esquema EThcD fornece espectros MS / MS mais informativos para localização de fosfosita inequívoca.
Uma caracterização detalhada dos locais de fosforilação é muito difícil, e a quantificação da fosforilação de proteínas por espectrometria de massa requer abordagens de padrão interno isotópico. Uma quantificação relativa pode ser obtida com uma variedade de tecnologias de marcação de isótopos diferenciais. Existem também vários métodos quantitativos de fosforilação de proteínas, incluindo imunoensaios de fluorescência, termoforese em microescala , FRET , TRF, polarização de fluorescência, extinção de fluorescência, mudança de mobilidade, detecção baseada em grânulos e formatos baseados em células.
Evolução
A fosforilação de proteínas é comum entre todos os clados da vida, incluindo todos os animais, plantas, fungos, bactérias e arquéias. As origens dos mecanismos de fosforilação de proteínas são ancestrais e divergem muito entre as diferentes espécies. Em eucariotos, estima-se que entre 30-65% de todas as proteínas podem ser fosforiladas, com dezenas ou mesmo centenas de milhares de locais de fosforilação distintos. Alguns locais de fosforilação parecem ter evoluído como interruptores "desligados" condicionais, bloqueando o local ativo de uma enzima, como na enzima metabólica procariótica isocitrato desidrogenase. No entanto, no caso de proteínas que devem ser fosforiladas para serem ativas, é menos claro como elas poderiam ter surgido de ancestrais não fosforilados. Foi demonstrado que um subconjunto de fosfositos de serina são frequentemente substituídos por resíduos ácidos, como aspartato e glutamato, entre diferentes espécies. Esses resíduos aniônicos podem interagir com resíduos catiônicos, como lisina e arginina, para formar pontes de sal , interações não covalentes estáveis que alteram a estrutura de uma proteína. Esses fosfositos frequentemente participam de pontes salinas, sugerindo que alguns locais de fosforilação evoluíram como interruptores "ligados" condicionais para pontes salinas, permitindo que essas proteínas adotassem uma conformação ativa apenas em resposta a um sinal específico.
Existem cerca de 600 proteínas quinases eucarióticas conhecidas, tornando-as uma das maiores famílias de genes. A maior parte da fosforilação é realizada por uma única superfamília de proteínas quinases que compartilham um domínio de quinase conservado. A fosforilação de proteínas é altamente conservada nas vias centrais para a sobrevivência celular, como a progressão do ciclo celular que depende de quinases dependentes de ciclina (CDKs), mas os locais de fosforilação individuais costumam ser flexíveis. Os alvos da fosforilação de CDK geralmente têm fosfositos em segmentos desordenados , que são encontrados em locais não idênticos, mesmo em espécies próximas. Por outro lado, os alvos de fosforilação de CDK em regiões estruturalmente definidas são mais altamente conservados. Enquanto a atividade de CDK é crítica para o crescimento e sobrevivência celular em todos os eucariotos, apenas muito poucos fosfositos mostram uma forte conservação de suas posições precisas. O posicionamento é provavelmente altamente importante para os fosfatos que regulam alostericamente a estrutura da proteína, mas muito mais flexível para os fosfatos que interagem com os domínios de ligação de fosfopeptídeos para recrutar proteínas regulatórias.
Comparações entre eucariotos e procariontes
A fosforilação de proteínas é uma modificação reversível pós-tradução de proteínas. Em eucariotos, as funções de fosforilação de proteínas na sinalização celular, expressão gênica e diferenciação. Também está envolvido na replicação do DNA durante o ciclo celular e nos mecanismos que lidam com os blocos de replicação induzidos pelo estresse. Comparados aos eucariotos, os procariotos usam quinases e fosfatases do tipo Hanks para a transdução de sinal. Ainda não está claro se a fosforilação de proteínas em bactérias também pode regular processos como reparo ou replicação de DNA.
Em comparação com a fosforilação de proteínas de procariotos, os estudos de fosforilação de proteínas em eucariotos de levedura para células humanas têm sido bastante extensos. Sabe-se que os eucariotos dependem da fosforilação do grupo hidroxila nas cadeias laterais da serina, treonina e tirosina para a sinalização celular. Estas são as principais modificações regulatórias pós-tradução em células eucarióticas, mas a fosforilação de proteínas de procariotos é menos intensamente estudada. Enquanto a serina, a treonina e a tirosina são fosforiladas em eucariotos, a histidina e o aspartato são fosforilados em procariotas, plantas e eucariotas não vegetais. Em bactérias, a fosforilação da histidina ocorre nos sistemas de fosfotransferase dependentes de fosfoenolpiruvato (PTSs), que estão envolvidos no processo de internalização, bem como na fosforilação de açúcares.
A fosforilação da proteína pela proteína quinase foi mostrada pela primeira vez em E. coli e Salmonella typhimurium, mas desde então foi demonstrada em muitas outras células bacterianas. Verificou-se que as bactérias usam a fosforilação de histidina e aspartato como modelo para transdução de sinalização bacteriana, mas nos últimos anos houve evidências de que a fosforilação de serina, treonina e tirosina também estão presentes em bactérias. Foi demonstrado que as bactérias carregam quinases e fosfatases semelhantes às de seus equivalentes eucarióticos, mas também desenvolveram quinases e fosfatases únicas não encontradas em eucariotos.
Patologia
A fosforilação anormal de proteínas tem sido implicada em uma série de doenças, notavelmente câncer , mas também doença de Alzheimer , doença de Parkinson e outras doenças degenerativas .
A proteína tau pertence a um grupo de proteínas associadas a microtúbulos (MAPs) que, entre várias coisas, ajudam a estabilizar os microtúbulos nas células, incluindo os neurônios. A atividade de associação e estabilização da proteína tau depende do seu estado fosforilado. Na doença de Alzheimer, devido a dobramentos incorretos e mudanças conformacionais anormais na estrutura da proteína tau, ela se torna ineficaz na ligação aos microtúbulos e, portanto, incapaz de manter a estrutura do citoesqueleto neural organizada durante os processos neurais; na verdade, a tau anormal inibe e interrompe a organização dos microtúbulos e desativa a tau normal dos microtúbulos para a fase citosólica. Os desdobramentos levam à agregação anormal em emaranhados fibrilares dentro dos neurônios, a marca registrada da doença de Alzheimer. Há uma quantidade adequada de que a proteína tau precisa ser fosforilada para funcionar, mas a hiperfosforilação da proteína tau é considerada uma das principais influências em sua incapacidade de associação. As fosfatases PP1, PP2A, PP2B e PP2C desfosforilam a proteína tau in vitro , e suas atividades foram reduzidas em áreas do cérebro em pacientes com Alzheimer. A fosfoproteína tau é três a quatro vezes hiperfosforilada em um paciente com Alzheimer em comparação com um indivíduo idoso não afetado. A doença de Alzheimer tau parece remover MAP1 e MAP2 (duas outras proteínas associadas principais) dos microtúbulos e esse efeito deletério é revertido quando a desfosforilação é realizada, evidenciando hiperfosforilação como a única causa da atividade incapacitante.
Mal de Parkinson
A α-sinucleína é uma proteína associada à doença de Parkinson. Essa proteína é codificada pelo gene PARRK1 e, em sua forma nativa, a α-sinucleína está envolvida na reciclagem das vesículas sinápticas que carregam os neurotransmissores e ocorre naturalmente na forma desdobrada. Níveis elevados de α-sinucleína são encontrados em pacientes com doença de Parkinson, e parece haver uma correlação positiva entre a quantidade da proteína α-sinucleína presente no paciente e a gravidade da doença.
A fosforilação do aminoácido Ser 129 na proteína α-sinucleína tem um efeito profundo na gravidade da doença. Parece haver correlação entre a concentração total de α-sinucleína (não fosforilada) e a gravidade dos sintomas em pacientes com doença de Parkinson. Pacientes saudáveis parecem ter níveis mais elevados de α-sinucleína não fosforilada do que pacientes com doença de Parkinson. Além disso, a medição das alterações na proporção das concentrações de α-Sinucleína fosforilada para α-Sinucleína não fosforilada dentro de um paciente pode ser um marcador potencial da progressão da doença
A fosforilação de Ser 129 está associada à agregação da proteína e danos adicionais ao sistema nervoso. Além disso, a agregação de α-sinucleína fosforilada pode ser aumentada se uma proteína-esqueleto pré-sináptica Sept4 estiver presente em quantidades insuficientes. É importante notar que a interação direta de α-Sinucleína com a proteína Sept4 inibe a fosforilação de Ser 129 . Observe, no entanto, que a fosforilação de Ser 129 pode ser observada sem agregação de sinucleína em condições de superexpressão