Microarray de oligonucleotídeo específico de metilação - Methylation specific oligonucleotide microarray
Microarray de oligonucleotídeo específico de metilação , também conhecido como microarray MSO , foi desenvolvido como uma técnica para mapear alterações de metilação epigenética no DNA de células cancerosas.
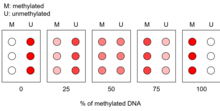
O processo geral começa com a modificação do DNA com bissulfito , especificamente para converter citosina não metilada em sítios CpG em uracila, enquanto deixa as citosinas metiladas intocadas. A região modificada do DNA de interesse é amplificada via PCR e, durante o processo, os uracilos são convertidos em timina. Os amplicons são marcados com um corante fluorescente e hibridizados com sondas oligonucleotídicas que são fixadas a uma lâmina de vidro. As sondas ligam-se diferencialmente aos resíduos de citosina e timina, o que em última análise permite a discriminação entre os locais CpG metilados e não metilados, respectivamente.
Uma curva de calibração é produzida e comparada com os resultados de microarray das amostras de DNA amplificadas. Isso permite uma quantificação geral da proporção de metilação presente na região de interesse.
Esta técnica de microarray foi desenvolvida por Tim Hui-Ming Huang e seu laboratório e foi publicada oficialmente em 2002.
Implicações para a pesquisa do câncer
As células cancerosas freqüentemente desenvolvem padrões de metilação atípicos , em locais CpG em promotores de genes supressores de tumor . Altos níveis de metilação em um promotor levam à regulação negativa dos genes correspondentes e são característicos da carcinogênese . É uma das alterações mais consistentes observadas nas células tumorais em estágio inicial. O microarray de oligonucleotídeo específico de metilação permite a detecção de alta resolução e alto rendimento de vários eventos de metilação em promotores de genes múltiplos. Portanto, esta técnica pode ser usada para detectar metilação aberrante em promotores supressores de tumor em um estágio inicial e tem sido usada em cânceres gástrico e de cólon e vários outros. Por permitir detectar a presença de metilação atípica em células cancerosas, também pode ser usado para revelar a principal causa da malignidade, se seu principal contribuidor são mutações nos cromossomos ou modificações epigenéticas, bem como quais níveis de transcrição de genes supressores de tumor são afetados. Um uso interessante desse microarray inclui a classificação específica de cânceres com base apenas nos padrões de metilação, como a diferenciação entre classes de leucemia , sugerindo que diferentes classes de câncer apresentam padrões de metilação relativamente únicos. Esta técnica também foi proposta para monitorar tratamentos de câncer que envolvem a modificação dos padrões de metilação em células cancerosas mutantes.
Referências
links externos
- Recursos, informações e protocolos específicos para análise de metilação de DNA
- Software para análise de metilação de DNA
| Este artigo é um esboço sobre Genética . Você pode ajudar a Wikipedia expandindo-a . |
| Este artigo sobre oncologia é um esboço . Você pode ajudar a Wikipedia expandindo-a . |