Amplificação de deslocamento múltiplo - Multiple displacement amplification
A amplificação de deslocamento múltiplo (MDA) é uma técnica de amplificação de DNA . Este método pode amplificar rapidamente pequenas quantidades de amostras de DNA a uma quantidade razoável para análise genômica. A reação começa pelo anelamento de primers hexâmeros aleatórios ao molde: a síntese de DNA é realizada por uma enzima de alta fidelidade , preferencialmente Φ29 DNA polimerase . Comparado com as técnicas convencionais de amplificação por PCR , o MDA não emprega iniciadores específicos para sequências, mas amplifica todo o DNA, gera produtos de tamanho maior com uma frequência de erro menor e funciona a uma temperatura constante. O MDA tem sido usado ativamente na amplificação do genoma completo (WGA) e é um método promissor para aplicação no sequenciamento do genoma de uma única célula e estudos genéticos baseados no sequenciamento.
Fundo
Muitos casos biológicos e forenses envolvendo análise genética requerem o sequenciamento de DNA de pequenas quantidades de amostra, como DNA de células isoladas não cultivadas ou vestígios de tecido coletado em cenas de crime. Os métodos convencionais de amplificação de DNA com base na Reação em Cadeia da Polimerase ( PCR ) requerem iniciadores oligonucleotídicos específicos de sequência e polimerase estável ao calor (geralmente Taq ) e podem ser usados para gerar quantidades significativas de DNA a partir de pequenas quantidades de DNA. No entanto, isso não é suficiente para técnicas modernas que usam análise de DNA baseada em sequenciamento. Portanto, um método não específico de sequência mais eficiente para amplificar pequenas quantidades de DNA é necessário, especialmente em estudos genômicos de uma única célula.
Materiais
Phi 29 DNA polimerase
O bacteriófago Φ29 DNA polimerase é uma enzima de alta processabilidade que pode produzir amplicons de DNA com mais de 70 pares de quilobases. A sua elevada fidelidade e 3'-5' actividade correctora reduz a taxa de erro de amplificação de 1 em cada 10 6 -10 7 bases em relação ao convencional de Taq polimerase com uma taxa de erro relatados de 1 em 9,000. A reação pode ser realizada em uma condição isotérmica moderada de 30 ° C e, portanto, não requer um termociclador . Ele tem sido usado ativamente na clonagem livre de células, que é o método enzimático de amplificação de DNA in vitro sem cultura de células e extração de DNA . O fragmento grande de Bst DNA polimerase também é usado em MDA, mas Ф29 é geralmente preferido devido ao seu rendimento de produto suficiente e atividade de revisão.
Hexamer primers
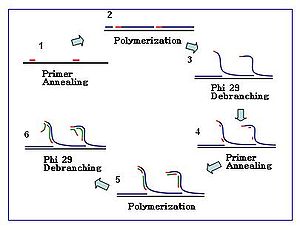
Os iniciadores hexâmeros são sequências compostas por seis nucleotídeos aleatórios . Para aplicações de MDA, esses primers são geralmente modificados com tiofosfato em sua extremidade 3 'para transmitir resistência à atividade de exonuclease 3'-5' da polimerase de DNA -29 . As reações de MDA começam com o emparelhamento de tais primers ao molde de DNA, seguido pelo alongamento da cadeia mediado pela polimerase. O número crescente de eventos de recozimento de primer acontecem ao longo da reação de amplificação.
Reação
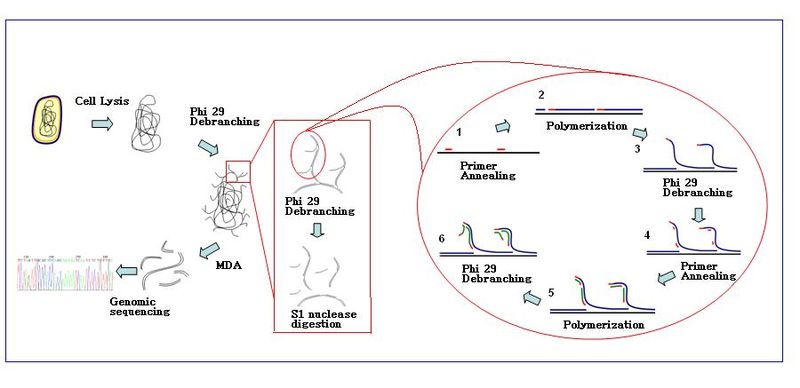
A reação de amplificação inicia quando múltiplos hexâmeros de primer se anelam ao molde. Quando a síntese de DNA prossegue para o próximo local de partida, a polimerase desloca a fita de DNA recém-produzida e continua seu alongamento. O deslocamento da fita gera um modelo de DNA de fita simples recém-sintetizado para que mais primers se recozam. O recozimento do primer e o deslocamento da fita no molde recém-sintetizado resultam em uma rede de DNA hiper-ramificada. A desramificação da sequência durante a amplificação resulta em um alto rendimento dos produtos. Para separar a rede de ramificação do DNA, as nucleases S1 são usadas para clivar os fragmentos em locais de deslocamento. Os entalhes sobre os fragmentos de ADN resultantes são reparados por ADN polimerase I .
Qualidade do produto
O MDA pode gerar 1–2 µg de DNA a partir de uma única célula com cobertura do genoma de até 99%. Os produtos também têm menor taxa de erro e tamanhos maiores em comparação com a amplificação Taq baseada em PCR .
Fluxo de trabalho geral do MDA:
- Preparação da amostra : as amostras são coletadas e diluídas no tampão de reação apropriado (livre de Ca 2+ e Mg 2+ ). As células são lisadas com tampão alcalino .
- Condição : A reação de MDA com a polimerase -29 é realizada a 30 ° C. A reação geralmente leva cerca de 2,5–3 horas.
- Fim da reação : Inativar as enzimas a 65 ° C antes da coleta dos produtos de DNA amplificados
- Os produtos de DNA podem ser purificados com o kit de purificação comercial.
Vantagens
O MDA gera rendimento suficiente de produtos de DNA. É uma ferramenta poderosa para amplificar moléculas de DNA de amostras, como microrganismos não cultivados ou células isoladas, em quantidade suficiente para estudos de sequenciamento . O grande tamanho dos produtos de DNA amplificados por MDA também fornece uma qualidade de amostra desejável para identificar o tamanho dos alelos de repetição polimórfica. Sua alta fidelidade também o torna confiável para ser usado na detecção de alelo de polimorfismo de nucleotídeo único (SNP). Devido ao deslocamento de sua fita durante a amplificação, o DNA amplificado tem cobertura suficiente das moléculas de DNA de origem, o que fornece um produto de alta qualidade para análise genômica. Os produtos das fitas deslocadas podem ser subsequentemente clonados em vetores para construir a biblioteca para reações de sequenciamento subsequentes .
Limitações
Desistência alélica (ADO)
ADO é definido como a não amplificação aleatória de um dos alelos presentes em uma amostra heterozigótica . Alguns estudos relataram que a taxa de ADO dos produtos de MDA é de 0–60%. Esta desvantagem diminui a precisão da genotipagem de uma única amostra e o diagnóstico incorreto em outras aplicações envolvidas com MDA. ADO parece ser independente dos tamanhos dos fragmentos e foi relatado que tem uma taxa semelhante em outras técnicas de célula única. As soluções possíveis são a utilização de diferentes condições de lise ou a realização de múltiplas rodadas de amplificações a partir dos produtos MDA diluídos, uma vez que foi relatado que a amplificação mediada por PCR de células em cultura produz taxas de ADO mais baixas.
Amplificação preferencial
'Amplificação preferencial' é a superamplificação de um dos alelos em comparação com o outro. A maioria dos estudos sobre MDA relatou esse problema. O viés de amplificação é atualmente considerado aleatório. Pode afetar a análise de pequenos trechos de DNA genômico na identificação de alelos Short Tandem Repeats (STR).
Interações primer-primer
A interação primer-primer independente do molde endógeno deve-se ao desenho aleatório dos primers hexâmeros. Uma solução possível é projetar iniciadores de hexanucleotídeos randomizados restritos que não hibridizam de forma cruzada.
Formulários
Sequenciamento do genoma de célula única
Células únicas de bactérias não cultivadas, arquéias e protistas, bem como partículas virais individuais e esporos de fungos únicos , foram sequenciadas com a ajuda do MDA.
A capacidade de sequenciar células individuais também é útil no combate a doenças humanas. Genomas de células embrionárias humanas individuais foram amplificados com sucesso para sequenciamento usando MDA, permitindo o diagnóstico genético pré-implantação (PGD): triagem de problemas de saúde genética em um embrião em estágio inicial antes da implantação . Doenças com propriedades heterogêneas , como câncer , também se beneficiam da capacidade de sequenciamento do genoma baseado em MDA para estudar mutações em células individuais.
Os produtos de MDA de uma única célula também foram usados com sucesso em experimentos de hibridização genômica comparativa de array , que geralmente requerem uma quantidade relativamente grande de DNA amplificado.
Imunoprecipitação da cromatina
A imunoprecipitação da cromatina resulta na produção de misturas complexas de fragmentos de DNA relativamente curtos, o que é desafiador para amplificar com MDA sem causar um viés na representação do fragmento. Um método para contornar este problema foi proposto, que é baseado na conversão dessas misturas em concatemers circulares usando ligação, seguido por Φ29 DNA polimerase mediada por MDA.
Análise forense
A quantidade de traços de amostras coletadas de cenas de crime pode ser amplificada pelo MDA para a quantidade que é suficiente para a análise forense de DNA, que é comumente usada na identificação de vítimas e suspeitos.