Paleopoliploidia - Paleopolyploidy
A paleopoliploidia é o resultado de duplicações do genoma que ocorreram há pelo menos vários milhões de anos (MYA). Tal evento poderia dobrar o genoma de uma única espécie ( autopoliploidia ) ou combinar os de duas espécies ( alopoliploidia ). Por causa da redundância funcional , os genes são rapidamente silenciados ou perdidos nos genomas duplicados. A maioria dos paleopoliploides, ao longo do tempo evolutivo, perdeu seu status poliploide por meio de um processo chamado diploidização e atualmente são considerados diploides, por exemplo, fermento de padeiro , Arabidopsis thaliana e talvez humanos .
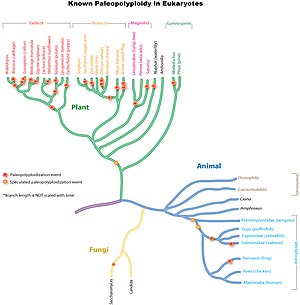
A paleopoliploidia é amplamente estudada em linhagens de plantas. Foi descoberto que quase todas as plantas com flores passaram por pelo menos uma rodada de duplicação do genoma em algum ponto durante sua história evolutiva. Duplicações do genoma antigo também são encontradas no ancestral dos vertebrados (que inclui a linhagem humana) perto da origem dos peixes ósseos , e outra na linhagem do caule dos peixes teleósteos. Evidências sugerem que a levedura de padeiro ( Saccharomyces cerevisiae ), que possui um genoma compacto, experimentou poliploidização durante sua história evolutiva.
O termo mesopoliplóide é algumas vezes usado para espécies que passaram por eventos de multiplicação do genoma completo (duplicação do genoma completo, triplificação do genoma completo, etc.) na história mais recente, como nos últimos 17 milhões de anos.
Eucariotos
Duplicações de genomas antigos são comuns em todas as linhagens eucarióticas , particularmente em plantas. Estudos sugerem que o ancestral comum de Poaceae , a família das gramíneas que inclui importantes espécies agrícolas como milho, arroz, trigo e cana-de-açúcar, compartilhou uma duplicação do genoma inteiro há cerca de 70 milhões de anos . Em linhagens de monocotiledôneas mais antigas, uma ou provavelmente várias rodadas de duplicações do genoma total ocorreram, as quais, entretanto, não foram compartilhadas com os eudicotiledôneas ancestrais . Outras duplicações independentes mais recentes do genoma total ocorreram nas linhagens que levam ao milho, cana-de-açúcar e trigo, mas não ao arroz, sorgo ou milheto.
Acredita-se que um evento de poliploidia há 160 milhões de anos tenha criado a linha ancestral que deu origem a todas as plantas com flores modernas. Esse evento de paleopoliploidia foi estudado pelo sequenciamento do genoma de uma antiga planta com flores, Amborella trichopoda .
Os eudicotes centrais também compartilhavam uma triplicação comum do genoma inteiro (paleo-hexaploidia), que se estima ter ocorrido após a divergência entre monocotiledôneas e eudicotiledôneas, mas antes da divergência de rósides e asterídeos . Muitas espécies de eudicot experimentaram duplicações ou triplicações adicionais do genoma inteiro. Por exemplo, a planta modelo Arabidopsis thaliana , a primeira planta a ter todo o seu genoma sequenciado, passou por pelo menos duas rodadas adicionais de duplicação do genoma inteiro desde a duplicação compartilhada pelos eudicotiledôneas centrais. O evento mais recente ocorreu antes da divergência das linhagens Arabidopsis e Brassica , cerca de 20 milhões de anos atrás a 45 milhões de anos atrás . Outros exemplos incluem os genomas de eudicot sequenciados de maçã, soja, tomate, algodão, etc.
Em comparação com as plantas, a paleopoliploidia é muito mais rara no reino animal. Foi identificado principalmente em anfíbios e peixes ósseos. Embora alguns estudos sugiram que uma ou mais duplicações comuns do genoma são compartilhadas por todos os vertebrados (incluindo humanos), a evidência não é tão forte como nos outros casos porque as duplicações, se existirem, aconteceram há muito tempo, e o assunto ainda está sob debate. A ideia de que os vertebrados compartilham uma duplicação do genoma inteiro comum é conhecida como a hipótese 2R . Muitos pesquisadores estão interessados na razão pela qual as linhagens animais, particularmente os mamíferos, tiveram tão menos duplicações do genoma completo do que as linhagens vegetais.
Uma paleopoliploidia bem suportada foi encontrada na levedura de padeiro ( Saccharomyces cerevisiae ), apesar de seu genoma pequeno e compacto (~ 13Mbp), após a divergência da levedura comum Kluyveromyces waltii . Por meio da racionalização do genoma, a levedura perdeu 90% do genoma duplicado ao longo do tempo evolutivo e agora é reconhecida como um organismo diplóide.
Método de detecção
Genes duplicados podem ser identificados por homologia de sequência no nível de DNA ou proteína. A paleopoliploidia pode ser identificada como uma duplicação maciça de genes de uma só vez, usando um relógio molecular . Para distinguir entre a duplicação do genoma inteiro e uma coleção de eventos de duplicação de gene único (mais comum) , as seguintes regras são frequentemente aplicadas:
- Genes duplicados estão localizados em grandes blocos duplicados. A duplicação de um único gene é um processo aleatório e tende a fazer genes duplicados espalhados por todo o genoma.
- Os blocos duplicados não são sobrepostos porque foram criados simultaneamente. A duplicação segmentar dentro do genoma pode cumprir a primeira regra; mas múltiplas duplicações segmentares independentes podem se sobrepor.
Em teoria, os dois genes duplicados deveriam ter a mesma "idade"; ou seja, a divergência da sequência deve ser igual entre os dois genes duplicados por paleopoliploidia ( homeólogos ). Taxa de substituição sinônima , Ks , é freqüentemente usada como um relógio molecular para determinar o tempo de duplicação do gene. Assim, a paleopoliploidia é identificada como um "pico" no gráfico de número duplicado vs. Ks (mostrado à direita).
Os eventos de duplicação que ocorreram há muito tempo na história de várias linhagens evolutivas podem ser difíceis de detectar por causa da diploidização subsequente (de tal forma que um poliploide começa a se comportar citogeneticamente como diplóide ao longo do tempo) conforme mutações e traduções de genes gradualmente fazem uma cópia de cada cromossomo ao contrário de sua contraparte. Isso geralmente resulta em uma baixa confiança para identificar uma paleopoliploidia muito antiga.
Importância evolutiva
Os eventos de paleopoliploidização levam a mudanças celulares massivas, incluindo a duplicação do material genético, mudanças na expressão gênica e aumento do tamanho das células. A perda de genes durante a diploidização não é completamente aleatória, mas fortemente selecionada. Genes de grandes famílias de genes são duplicados. Por outro lado, os genes individuais não são duplicados. No geral, a paleopoliploidia pode ter efeitos evolutivos de curto e longo prazo na aptidão de um organismo no ambiente natural.
- Evolução fenotípica aprimorada
- A duplicação do genoma inteiro pode aumentar as taxas e eficiência pelas quais os organismos adquirem novas características biológicas. No entanto, um teste dessa hipótese, que comparou as taxas evolutivas em inovação nos primeiros peixes teleósteos (com genomas duplicados) e nos primeiros peixes holosteanos (sem genomas duplicados), encontrou pouca diferença entre os dois.
- Diversidade de genoma
- A duplicação do genoma forneceu ao organismo alelos redundantes que podem evoluir livremente com pouca pressão de seleção. Os genes duplicados podem sofrer neofuncionalização ou subfuncionalização, o que pode ajudar o organismo a se adaptar ao novo ambiente ou sobreviver a diferentes condições de estresse.
- Vigor híbrido
- Os poliplóides costumam ter células maiores e órgãos ainda maiores. Muitas safras importantes, incluindo trigo, milho e algodão , são paleopoliplóides que foram selecionados para domesticação por povos antigos.
- Especiação
- Foi sugerido que muitos eventos de poliploidização criaram novas espécies, por meio de um ganho de características adaptativas ou por incompatibilidade sexual com suas contrapartes diplóides. Um exemplo seria a recente especiação do alopoliploide Spartina - S. anglica ; a planta poliplóide é tão bem-sucedida que é listada como uma espécie invasora em muitas regiões.
Alopoliploidia e autopoliploidia
Existem duas divisões principais de poliploidia , alopoliploidia e autopoliploidia. Os alopoliploides surgem como resultado da hibridização de duas espécies relacionadas, enquanto os autopoliplóides surgem da duplicação do genoma de uma espécie como resultado da hibridização de dois pais coespecíficos ou duplicação somática no tecido reprodutivo de um dos pais. Acredita-se que as espécies alopoliploides sejam muito mais prevalentes na natureza, possivelmente porque os alopoliploides herdam genomas diferentes, resultando em maior heterozigosidade e, portanto, maior aptidão. Esses diferentes genomas resultam em uma maior probabilidade de grandes reorganizações genômicas, que podem ser deletérias ou vantajosas. A autopoliploidia, no entanto, é geralmente considerada um processo neutro, embora tenha sido levantada a hipótese de que a autopoliploidia pode servir como um mecanismo útil para induzir a especiação e, portanto, auxiliar na capacidade de um organismo colonizar rapidamente em novos habitats sem sofrer período intensivo e caro de reorganização genômica experimentado por espécies alopoliplóides. Uma fonte comum de autopoliploidia em plantas provém de "flores perfeitas", que são capazes de autopolinização ou "autofecundação". Isso, junto com erros na meiose que levam à aneuploidia , pode criar um ambiente onde a autopoliploidia é muito provável. Este fato pode ser explorado em um ambiente de laboratório usando colchicina para inibir a segregação cromossômica durante a meiose, criando plantas autopoliplóides sintéticas.
Após eventos de poliploidia, há vários destinos possíveis para genes duplicados ; ambas as cópias podem ser retidas como genes funcionais, a mudança na função do gene pode ocorrer em uma ou ambas as cópias, o silenciamento do gene pode mascarar uma ou ambas as cópias, ou pode ocorrer a perda completa do gene. Os eventos de poliploidia resultarão em níveis mais altos de heterozigosidade e, com o tempo, podem levar a um aumento no número total de genes funcionais no genoma. Conforme o tempo passa após um evento de duplicação do genoma, muitos genes mudarão de função como resultado de qualquer mudança na função do gene duplicado para espécies alo e autopoliplóides, ou haverá mudanças na expressão gênica causada por rearranjos genômicos induzidos pela duplicação do genoma em alopoliploides . Quando ambas as cópias de um gene são retidas e, portanto, o número de cópias dobrado, há uma chance de que haja um aumento proporcional na expressão desse gene, resultando na produção de duas vezes mais transcrição de mRNA . Também existe a possibilidade de que a transcrição de um gene duplicado seja regulada negativamente, resultando em um aumento de menos de duas vezes na transcrição desse gene, ou que o evento de duplicação produza um aumento de mais de duas vezes na transcrição. Em uma espécie, Glycine dolichocarpa (um parente próximo da soja , Glycine max ), foi observado que após uma duplicação do genoma há cerca de 500.000 anos, houve um aumento de 1,4 vezes na transcrição, indicando que houve uma diminuição proporcional na transcrição em relação ao número de cópias do gene após o evento de duplicação.
Vertebrados como paleopoliplóides
A hipótese da paleopoliploidia em vertebrados surgiu já na década de 1970, proposta pelo biólogo Susumu Ohno . Ele argumentou que o genoma dos vertebrados não poderia atingir sua complexidade sem duplicações do genoma inteiro em grande escala. A hipótese das "duas rodadas de duplicação do genoma" (hipótese 2R ) surgiu e ganhou popularidade, especialmente entre os biólogos do desenvolvimento.
Alguns pesquisadores questionaram a hipótese 2R porque ela prevê que os genomas de vertebrados devem ter uma proporção de genes de 4: 1 em comparação com os genomas de invertebrados, e isso não é apoiado pelos resultados dos 48 projetos de genoma de vertebrados disponíveis em meados de 2011. Por exemplo, o genoma humano consiste em ~ 21.000 genes codificadores de proteínas de acordo com as contagens de junho de 2011 nos centros de análise de genoma da UCSC e Ensembl, enquanto o tamanho médio do genoma de um invertebrado é de cerca de 15.000 genes. A sequência do genoma do anfioxus forneceu suporte para a hipótese de duas rodadas de duplicação do genoma inteiro, seguido pela perda de cópias duplicadas da maioria dos genes. Argumentos adicionais contra 2R foram baseados na falta de topologia de árvore (AB) (CD) entre quatro membros de uma família de genes em vertebrados. No entanto, se as duas duplicações do genoma ocorressem juntas, não esperaríamos encontrar essa topologia. Um estudo recente gerou o mapa genético da lampreia marinha , que forneceu um forte suporte para a hipótese de que uma única duplicação do genoma completo ocorreu na linhagem de vertebrados basais, precedida e seguida por várias duplicações segmentais evolutivamente independentes que ocorreram durante a evolução dos cordados.
Veja também
Referências
Leitura adicional
- Adams KL, Wendel JF (abril de 2005). "Poliploidia e evolução do genoma nas plantas". Opinião atual em Biologia Vegetal . 8 (2): 135–41. doi : 10.1016 / j.pbi.2005.01.001 . PMID 15752992 .
- Cui L, Wall PK, Leebens-Mack JH, Lindsay BG, Soltis DE, Doyle JJ, et al. (Junho de 2006). Duplicações generalizadas do genoma ao longo da história das plantas com flores " . Genome Research . 16 (6): 738–49. doi : 10.1101 / gr.4825606 . PMC 1479859 . PMID 16702410 .
- Comai L (novembro de 2005). “As vantagens e desvantagens de ser poliplóide”. Nature Reviews. Genética . 6 (11): 836–46. doi : 10.1038 / nrg1711 . PMID 16304599 . S2CID 3329282 .
- Otto SP, Whitton J (2000). “Incidência e evolução poliplóide”. Revisão Anual de Genética . 34 (1): 401–437. CiteSeerX 10.1.1.323.1059 . doi : 10.1146 / annurev.genet.34.1.401 . PMID 11092833 .
- Makalowski W (maio de 2001). "Somos poliplóides? Uma breve história de uma hipótese" . Genome Research . 11 (5): 667–70. doi : 10.1101 / gr.188801 . PMID 11337465 .
- Kellis M, Birren BW, Lander ES (abril de 2004). "Prova e análise evolutiva da duplicação do genoma antigo na levedura Saccharomyces cerevisiae". Nature . 428 (6983): 617–24. Bibcode : 2004Natur.428..617K . doi : 10.1038 / nature02424 . PMID 15004568 . S2CID 4422074 .