Computação de imagens médicas - Medical image computing
A computação de imagens médicas (MIC) é um campo interdisciplinar na interseção da ciência da computação , engenharia da informação , engenharia elétrica , física , matemática e medicina . Este campo desenvolve métodos computacionais e matemáticos para resolver problemas relativos a imagens médicas e seu uso para pesquisas biomédicas e cuidados clínicos.
O principal objetivo do MIC é extrair informações ou conhecimento clinicamente relevantes de imagens médicas. Embora intimamente relacionado ao campo da imagem médica , o MIC se concentra na análise computacional das imagens, não em sua aquisição. Os métodos podem ser agrupadas em várias categorias amplas: segmentação de imagens , registro de imagem , modelagem fisiológica baseado em imagem , e outros.
Formulários de dados
A computação de imagens médicas normalmente opera em dados amostrados uniformemente com espaçamento espacial xyz regular (imagens em 2D e volumes em 3D, genericamente chamados de imagens). Em cada ponto de amostra, os dados são comumente representados em forma integral , como curto com sinal e sem sinal (16 bits), embora formas de char sem sinal (8 bits) a flutuante de 32 bits não sejam incomuns. O significado particular dos dados no ponto de amostra depende da modalidade: por exemplo, uma aquisição de TC coleta valores de radiodensidade, enquanto uma aquisição de MRI pode coletar imagens ponderadas em T1 ou T2 . As aquisições longitudinais que variam no tempo podem ou não adquirir imagens com intervalos regulares de tempo. Imagens em leque devido a modalidades como ultrassom de matriz curva também são comuns e requerem diferentes técnicas representacionais e algorítmicas para serem processadas. Outras formas de dados incluem imagens distorcidas devido à inclinação do gantry durante a aquisição; e malhas não estruturadas , como as formas hexaédrica e tetraédrica, que são usadas em análises biomecânicas avançadas (por exemplo, deformação de tecidos, transporte vascular, implantes ósseos).
Segmentação
A segmentação é o processo de particionar uma imagem em diferentes segmentos significativos. Em imagens médicas, esses segmentos geralmente correspondem a diferentes classes de tecidos, órgãos , patologias ou outras estruturas biologicamente relevantes. A segmentação de imagens médicas é dificultada por baixo contraste, ruído e outras ambigüidades de imagem. Embora existam muitas técnicas de visão por computador para segmentação de imagens , algumas foram adaptadas especificamente para a computação de imagens médicas. Abaixo está uma amostra das técnicas neste campo; a implementação depende da experiência que os médicos podem fornecer.
- Segmentação baseada em Atlas : para muitas aplicações, um especialista clínico pode rotular manualmente várias imagens; segmentar imagens invisíveis é uma questão de extrapolar a partir dessas imagens de treinamento rotuladas manualmente. Os métodos desse estilo são normalmente chamados de métodos de segmentação baseados em atlas. Os métodos de atlas paramétricos geralmente combinam essas imagens de treinamento em uma única imagem de atlas, enquanto os métodos de atlas não paramétricos geralmente usam todas as imagens de treinamento separadamente. Os métodos baseados em Atlas geralmente requerem o uso de registro de imagem para alinhar a imagem ou imagens do atlas a uma nova imagem não vista.
- Segmentação baseada em forma : Muitos métodos parametrizam uma forma de template para uma determinada estrutura, freqüentemente contando com pontos de controle ao longo do limite. A forma inteira é então deformada para corresponder a uma nova imagem. Duas das técnicas mais comuns baseadas em forma são os Modelos de Forma Ativos e os Modelos de Aparência Ativos. Esses métodos foram muito influentes e deram origem a modelos semelhantes.
- Segmentação baseada em imagem : Alguns métodos iniciam um modelo e refinam sua forma de acordo com os dados da imagem enquanto minimizam as medidas de erro integral, como o modelo de contorno ativo e suas variações.
- Segmentação interativa : os métodos interativos são úteis quando os médicos podem fornecer algumas informações, como uma região de semente ou um esboço da região a ser segmentada. Um algoritmo pode, então, refinar iterativamente essa segmentação, com ou sem orientação do médico. A segmentação manual, usando ferramentas como um pincel para definir explicitamente a classe de tecido de cada pixel, continua sendo o padrão ouro para muitas aplicações de imagem. Recentemente, princípios da teoria de controle de feedback foram incorporados à segmentação, o que dá ao usuário muito maior flexibilidade e permite a correção automática de erros.
- Segmentação subjetiva de superfície: Este método é baseado na ideia de evolução da função de segmentação que é governada por um modelo de difusão-advecção. Para segmentar um objeto, é necessária uma semente de segmentação (que é o ponto de partida que determina a posição aproximada do objeto na imagem). Consequentemente, uma função de segmentação inicial é construída. A ideia por trás do método subjetivo de superfície é que a posição da semente é o principal fator que determina a forma dessa função de segmentação.
No entanto, existem algumas outras classificações de métodos de segmentação de imagens que são semelhantes às categorias acima. Além disso, podemos classificar outro grupo como "Híbrido" que se baseia na combinação de métodos.
Cadastro
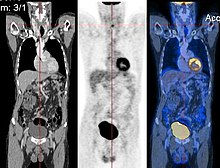
O registro de imagens é um processo que busca o alinhamento correto das imagens. No caso mais simples, duas imagens são alinhadas. Normalmente, uma imagem é tratada como a imagem de destino e a outra é tratada como uma imagem de origem; a imagem de origem é transformada para corresponder à imagem de destino. O procedimento de otimização atualiza a transformação da imagem de origem com base em um valor de similaridade que avalia a qualidade atual do alinhamento. Este procedimento iterativo é repetido até que um ótimo (local) seja encontrado. Um exemplo é o registro de imagens de TC e PET para combinar informações estruturais e metabólicas (veja a figura).
O registro de imagens é usado em uma variedade de aplicações médicas:
- Estudando mudanças temporais. Os estudos longitudinais adquirem imagens ao longo de vários meses ou anos para estudar processos de longo prazo, como a progressão da doença. As séries temporais correspondem às imagens adquiridas na mesma sessão (segundos ou minutos). Eles podem ser usados para estudar processos cognitivos, deformações cardíacas e respiração.
- Combinando informações complementares de diferentes modalidades de imagem . Um exemplo é a fusão de informações anatômicas e funcionais. Como o tamanho e a forma das estruturas variam entre as modalidades, é mais desafiador avaliar a qualidade do alinhamento. Isso levou ao uso de medidas de similaridade , como informações mútuas .
- Caracterizando uma população de sujeitos. Em contraste com o registro intra-sujeito, um mapeamento um para um pode não existir entre os sujeitos, dependendo da variabilidade estrutural do órgão de interesse. O registro intersujeito é necessário para a construção de atlas em anatomia computacional . Aqui, o objetivo é modelar estatisticamente a anatomia dos órgãos em todos os assuntos.
- Cirurgia assistida por computador . Em imagens pré-operatórias de cirurgia assistida por computador, como tomografia computadorizada ou ressonância magnética, são registradas em imagens intra-operatórias ou sistemas de rastreamento para facilitar a orientação ou navegação da imagem.
Existem várias considerações importantes ao realizar o registro da imagem:
- O modelo de transformação . As escolhas comuns são modelos de transformação rígidos , afins e deformáveis . Modelos de spline B e placa fina são comumente usados para campos de transformação parametrizados. Os campos de deformação não paramétricos ou densos carregam um vetor de deslocamento em cada localização da grade; isso requer restrições de regularização adicionais . Uma classe específica de campos de deformação são os difeomorfismos , que são transformações invertíveis com um inverso suave.
- A métrica de similaridade. Uma função de distância ou similaridade é usada para quantificar a qualidade do registro. Essa semelhança pode ser calculada nas imagens originais ou nas características extraídas das imagens. As medidas de similaridade comuns são soma das distâncias quadradas (SSD), coeficiente de correlação e informações mútuas . A escolha da medida de similaridade depende se as imagens são da mesma modalidade; o ruído de aquisição também pode desempenhar um papel nesta decisão. Por exemplo, SSD é a medida de similaridade ideal para imagens da mesma modalidade com ruído gaussiano . No entanto, as estatísticas de imagem em ultrassom são significativamente diferentes do ruído gaussiano, levando à introdução de medidas de similaridade específicas de ultrassom. O registro multimodal requer uma medida de similaridade mais sofisticada; alternativamente, uma representação de imagem diferente pode ser usada, como representações estruturais ou registro de anatomia adjacente.
- O procedimento de otimização . A otimização contínua ou discreta é executada. Para otimização contínua, técnicas de otimização baseadas em gradiente são aplicadas para melhorar a velocidade de convergência.
Visualização
A visualização desempenha vários papéis importantes na computação de imagens médicas. Métodos de visualização científica são usados para entender e comunicar sobre imagens médicas, que são inerentemente espaço-temporais. A visualização e a análise de dados são usadas em formulários de dados não estruturados , por exemplo, ao avaliar medidas estatísticas derivadas durante o processamento algorítmico. A interação direta com os dados, um recurso-chave do processo de visualização, é usada para realizar consultas visuais sobre os dados, fazer anotações em imagens, orientar os processos de segmentação e registro e controlar a representação visual dos dados (controlando as propriedades de renderização de iluminação e os parâmetros de visualização). A visualização é usada tanto para a exploração inicial quanto para transmitir os resultados intermediários e finais das análises.
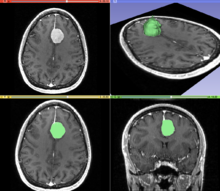
A figura "Visualização de imagens médicas" ilustra vários tipos de visualização: 1. a exibição de seções transversais como imagens em escala de cinza; 2. visualizações reformatadas de imagens em escala de cinza (a visualização sagital neste exemplo tem uma orientação diferente da direção original da aquisição da imagem; e 3. Uma renderização de volume 3D dos mesmos dados. A lesão nodular é claramente visível nas diferentes apresentações e foi anotado com uma linha branca.
Atlas
As imagens médicas podem variar significativamente entre os indivíduos devido ao fato de as pessoas possuírem órgãos de diferentes formas e tamanhos. Portanto, representar imagens médicas para explicar essa variabilidade é crucial. Uma abordagem popular para representar imagens médicas é por meio do uso de um ou mais atlas. Aqui, um atlas se refere a um modelo específico para uma população de imagens com parâmetros que são aprendidos a partir de um conjunto de dados de treinamento.
O exemplo mais simples de atlas é uma imagem de intensidade média, comumente chamada de gabarito. No entanto, um atlas também pode incluir informações mais ricas, como estatísticas de imagens locais e a probabilidade de que um determinado local espacial tenha um determinado rótulo. Novas imagens médicas, que não são utilizadas durante o treinamento, podem ser mapeadas em um atlas, que foi adaptado para a aplicação específica, como segmentação e análise de grupo . Mapear uma imagem para um atlas geralmente envolve o registro da imagem e do atlas. Essa deformação pode ser usada para lidar com a variabilidade em imagens médicas.
Template único
A abordagem mais simples é modelar imagens médicas como versões deformadas de uma única imagem de modelo. Por exemplo, varreduras cerebrais anatômicas de ressonância magnética são frequentemente mapeadas para o modelo MNI para representar todas as varreduras cerebrais em coordenadas comuns. A principal desvantagem de uma abordagem de modelo único é que, se houver diferenças significativas entre o modelo e uma determinada imagem de teste, pode não haver uma boa maneira de mapear um no outro. Por exemplo, uma ressonância magnética cerebral anatômica de um paciente com anormalidades cerebrais graves (ou seja, um tumor ou procedimento cirúrgico) pode não ser mapeada facilmente para o modelo MNI.
Múltiplos modelos
Em vez de depender de um único modelo, vários modelos podem ser usados. A ideia é representar uma imagem como uma versão deformada de um dos modelos. Por exemplo, pode haver um modelo para uma população saudável e um modelo para uma população doente. No entanto, em muitos aplicativos, não está claro quantos modelos são necessários. Uma maneira simples, embora computacionalmente cara de lidar com isso, é fazer com que cada imagem em um conjunto de dados de treinamento seja uma imagem de modelo e, assim, cada nova imagem encontrada é comparada com cada imagem no conjunto de dados de treinamento. Uma abordagem mais recente encontra automaticamente o número de modelos necessários.
Análise estatística
Os métodos estatísticos combinam o campo da imagem médica com a moderna visão computacional , aprendizado de máquina e reconhecimento de padrões . Ao longo da última década, vários grandes conjuntos de dados foram disponibilizados publicamente (ver por exemplo ADNI, 1000 Functional Connectomes Project), em parte devido à colaboração entre vários institutos e centros de pesquisa. Esse aumento no tamanho dos dados exige novos algoritmos que podem explorar e detectar mudanças sutis nas imagens para tratar de questões clínicas. Essas questões clínicas são muito diversas e incluem análise de grupo, biomarcadores de imagem, fenotipagem de doenças e estudos longitudinais.
Análise de grupo
Na Análise de Grupo, o objetivo é detectar e quantificar anormalidades induzidas por uma doença, comparando as imagens de duas ou mais coortes. Normalmente, uma dessas coortes consiste em indivíduos normais (controle) e a outra consiste em pacientes anormais. A variação causada pela doença pode se manifestar como deformação anormal da anatomia (consulte morfometria baseada em Voxel ). Por exemplo, o encolhimento de tecidos subcorticais como o hipocampo no cérebro pode estar relacionado à doença de Alzheimer . Além disso, as alterações na atividade bioquímica (funcional) podem ser observadas usando modalidades de imagem como a tomografia por emissão de pósitrons .
A comparação entre os grupos geralmente é realizada no nível do voxel . Portanto, o pipeline de pré-processamento mais popular, particularmente em neuroimagem , transforma todas as imagens em um conjunto de dados em um quadro de coordenadas comum via ( Registro de imagens médicas ) para manter a correspondência entre os voxels. Dada essa correspondência voxel, o método Frequentista mais comum é extrair uma estatística para cada voxel (por exemplo, a intensidade média do voxel para cada grupo) e realizar testes de hipótese estatística para avaliar se uma hipótese nula é ou não suportada. A hipótese nula normalmente assume que as duas coortes são tiradas da mesma distribuição e, portanto, devem ter as mesmas propriedades estatísticas (por exemplo, os valores médios de dois grupos são iguais para um determinado voxel). Uma vez que as imagens médicas contêm um grande número de voxels, o problema da comparação múltipla precisa ser abordado. Existem também abordagens bayesianas para lidar com o problema de análise de grupo.
Classificação
Embora a análise de grupo possa quantificar os efeitos gerais de uma patologia em uma anatomia e função, ela não fornece medidas em nível de sujeito e, portanto, não pode ser usada como biomarcadores para diagnóstico (consulte Biomarcadores de imagem). Os médicos, por outro lado, estão frequentemente interessados no diagnóstico precoce da patologia (isto é, classificação) e em aprender a progressão de uma doença (isto é, regressão). Do ponto de vista metodológico, as técnicas atuais variam da aplicação de algoritmos de aprendizado de máquina padrão a conjuntos de dados de imagens médicas (por exemplo, Support Vector Machine ), ao desenvolvimento de novas abordagens adaptadas às necessidades do campo. As principais dificuldades são as seguintes:
- Tamanho de amostra pequeno ( Curse of Dimensionality ): um grande conjunto de dados de imagens médicas contém centenas a milhares de imagens, enquanto o número de voxels em uma imagem volumétrica típica pode facilmente ultrapassar milhões. Uma solução para esse problema é reduzir o número de recursos em um sentido informativo (ver redução de dimensionalidade ). Várias abordagens não supervisionadas e semi / supervisionadas têm sido propostas para resolver esse problema.
- Interpretabilidade: uma boa precisão de generalização nem sempre é o objetivo principal, pois os médicos gostariam de compreender quais partes da anatomia são afetadas pela doença. Portanto, a interpretabilidade dos resultados é muito importante; métodos que ignoram a estrutura da imagem não são favorecidos. Métodos alternativos baseados na seleção de características foram propostos.
Clustering
Os métodos de classificação de padrões baseados em imagens normalmente assumem que os efeitos neurológicos de uma doença são distintos e bem definidos. Isso pode nem sempre ser o caso. Para uma série de condições médicas, as populações de pacientes são altamente heterogêneas e a categorização em subcondições não foi estabelecida. Além disso, algumas doenças (por exemplo, transtorno do espectro do autismo (TEA), esquizofrenia , comprometimento cognitivo leve (MCI)) podem ser caracterizadas por um espectro contínuo ou quase contínuo de comprometimento cognitivo leve a alterações patológicas muito pronunciadas. Para facilitar a análise baseada em imagens de distúrbios heterogêneos, foram desenvolvidas alternativas metodológicas para a classificação de padrões. Essas técnicas emprestam ideias de agrupamento de alta dimensão e regressão de padrão de alta dimensão para agrupar uma determinada população em subpopulações homogêneas. O objetivo é fornecer uma melhor compreensão quantitativa da doença em cada subpopulação.
Análise de forma
A Análise de Forma é a área da Computação de Imagens Médicas que estuda propriedades geométricas de estruturas obtidas a partir de diferentes modalidades de imagem . A análise de formas tornou-se recentemente de interesse crescente para a comunidade médica devido ao seu potencial para localizar com precisão as mudanças morfológicas entre diferentes populações de estruturas, ou seja, saudáveis versus patológicas, mulheres versus homens, jovens versus idosos. A Análise de formas inclui duas etapas principais: correspondência de formas e análise estatística.
- Correspondência de formas é a metodologia que calcula localizações correspondentes entre formas geométricas representadas por malhas triangulares, contornos, conjuntos de pontos ou imagens volumétricas. Obviamente, a definição de correspondência influenciará diretamente a análise. Entre as diferentes opções para estruturas de correspondência, podemos encontrar: correspondência anatômica, pontos de referência manuais, correspondência funcional (ou seja, no locus morfométrico do cérebro responsável pela mesma funcionalidade neuronal), correspondência de geometria, (para volumes de imagem) similaridade de intensidade, etc. Algumas abordagens, por exemplo, espectral análise de forma, não requer correspondência, mas compare os descritores de forma diretamente.
- A análise estatística fornecerá medidas de mudança estrutural em locais correspondentes.
Estudos longitudinais
Em estudos longitudinais, a imagem da mesma pessoa é repetida várias vezes. Essas informações podem ser incorporadas tanto na análise de imagens , quanto na modelagem estatística.
- No processamento longitudinal de imagens, os métodos de segmentação e análise de pontos de tempo individuais são informados e regularizados com informações comuns, geralmente de um modelo dentro do assunto. Essa regularização foi projetada para reduzir o ruído de medição e, portanto, ajuda a aumentar a sensibilidade e o poder estatístico. Ao mesmo tempo, a super regularização precisa ser evitada, para que os tamanhos de efeito permaneçam estáveis. A regularização intensa, por exemplo, pode levar a uma excelente confiabilidade teste-reteste, mas limita a capacidade de detectar quaisquer mudanças e diferenças reais entre os grupos. Freqüentemente, é necessário buscar uma compensação que otimiza a redução de ruído ao custo de uma perda limitada de tamanho de efeito. Outro desafio comum no processamento de imagens longitudinais é a, muitas vezes não intencional, introdução de viés de processamento. Quando, por exemplo, as imagens de acompanhamento são registradas e reamostradas para a imagem de linha de base, os artefatos de interpolação são introduzidos apenas nas imagens de acompanhamento e não na linha de base. Esse artefato pode causar efeitos espúrios (geralmente um viés para superestimar a mudança longitudinal e, portanto, subestimar o tamanho da amostra necessária). Portanto, é essencial que todos os pontos de tempo sejam tratados exatamente da mesma forma para evitar qualquer viés de processamento.
- O pós-processamento e a análise estatística de dados longitudinais geralmente requerem ferramentas estatísticas dedicadas, como ANOVA de medida repetida ou os modelos lineares de efeitos mistos mais poderosos. Além disso, é vantajoso considerar a distribuição espacial do sinal. Por exemplo, as medições da espessura cortical mostrarão uma correlação dentro do sujeito ao longo do tempo e também dentro de uma vizinhança na superfície cortical - um fato que pode ser usado para aumentar o poder estatístico. Além disso, a análise de tempo até o evento (também conhecida como sobrevivência) é frequentemente empregada para analisar dados longitudinais e determinar preditores significativos.
Modelagem fisiológica baseada em imagem
Tradicionalmente, a computação de imagens médicas aborda a quantificação e a fusão de informações estruturais ou funcionais disponíveis no ponto e no momento da aquisição da imagem. Nesse sentido, pode ser visto como uma detecção quantitativa dos processos anatômicos, físicos ou fisiológicos subjacentes. No entanto, nos últimos anos, tem havido um interesse crescente na avaliação preditiva da doença ou do curso da terapia. A modelagem baseada em imagens, seja de natureza biomecânica ou fisiológica, pode, portanto, estender as possibilidades da computação de imagens de um ângulo descritivo para um preditivo.
De acordo com o roteiro de pesquisa STEP, o Virtual Fisiológico Humano (VPH) é um arcabouço metodológico e tecnológico que, uma vez estabelecido, permitirá a investigação do corpo humano como um único sistema complexo. Subjacente ao conceito de VPH, a União Internacional para Ciências Fisiológicas (IUPS) tem patrocinado o Projeto IUPS Physiome por mais de uma década. Este é um esforço de domínio público mundial para fornecer uma estrutura computacional para a compreensão da fisiologia humana. Seu objetivo é desenvolver modelos integrativos em todos os níveis de organização biológica, desde genes até organismos inteiros, por meio de redes regulatórias de genes, vias de proteínas, funções integrativas de células e relações entre tecido e estrutura / função de todo o órgão. Tal abordagem visa transformar a prática atual da medicina e sustenta uma nova era da medicina computacional.
Nesse contexto, a imagiologia médica e a computação de imagens desempenham um papel cada vez mais importante, pois fornecem sistemas e métodos para imagear, quantificar e fundir informações estruturais e funcionais sobre o ser humano in vivo. Essas duas grandes áreas de pesquisa incluem a transformação de modelos computacionais genéricos para representar assuntos específicos, abrindo caminho para modelos computacionais personalizados. A individualização de modelos computacionais genéricos por meio de imagens pode ser realizada em três direções complementares:
- definição do domínio computacional específico do assunto (anatomia) e subdomínios relacionados (tipos de tecido);
- definição de limites e condições iniciais de imagens (dinâmicas e / ou funcionais); e
- caracterização das propriedades estruturais e funcionais do tecido.
Além disso, a imagem também desempenha um papel fundamental na avaliação e validação de tais modelos, tanto em humanos quanto em modelos animais, e na tradução de modelos para o ambiente clínico com aplicações diagnósticas e terapêuticas. Neste contexto específico, imagens moleculares, biológicas e pré-clínicas fornecem dados adicionais e compreensão da estrutura básica e função em moléculas, células, tecidos e modelos animais que podem ser transferidos para a fisiologia humana quando apropriado.
As aplicações de modelos VPH / Physiome baseados em imagens em domínios básicos e clínicos são vastas. Em termos gerais, eles prometem se tornar novas técnicas de imagem virtual . Efetivamente mais, muitas vezes não observáveis, os parâmetros serão visualizados in silico com base na integração de imagens multimodais observáveis, mas às vezes esparsas e inconsistentes e medições fisiológicas. Os modelos computacionais servirão para gerar a interpretação das medições de uma forma compatível com as leis biofísicas, bioquímicas ou biológicas subjacentes dos processos fisiológicos ou fisiopatológicos sob investigação. Em última análise, tais ferramentas e sistemas investigativos ajudarão nossa compreensão dos processos da doença, da história natural da evolução da doença e da influência dos procedimentos terapêuticos farmacológicos e / ou intervencionistas no curso de uma doença.
A fertilização cruzada entre a imagem e a modelagem vai além da interpretação das medições de uma forma consistente com a fisiologia. A modelagem específica do paciente baseada em imagens, combinada com modelos de dispositivos médicos e terapias farmacológicas, abre o caminho para imagens preditivas em que se será capaz de compreender, planejar e otimizar tais intervenções in silico .
Métodos matemáticos em imagens médicas
Vários métodos matemáticos sofisticados foram incorporados à imagem médica e já foram implementados em vários pacotes de software. Isso inclui abordagens baseadas em equações diferenciais parciais (PDEs) e fluxos orientados por curvatura para aprimoramento, segmentação e registro. Uma vez que eles empregam PDEs, os métodos são passíveis de paralelização e implementação em GPGPUs. Várias dessas técnicas foram inspiradas em ideias de controle ideal . Conseqüentemente, muito recentemente as idéias de controle fizeram seu caminho para os métodos interativos, especialmente a segmentação. Além disso, devido ao ruído e à necessidade de técnicas de estimativa estatística para imagens que mudam mais dinamicamente, o filtro de Kalman e o filtro de partículas começaram a ser usados. Um levantamento desses métodos com uma extensa lista de referências pode ser encontrado em.
Computação específica de modalidade
Algumas modalidades de imagem fornecem informações muito especializadas. As imagens resultantes não podem ser tratadas como imagens escalares regulares e dão origem a novas subáreas da Computação de Imagens Médicas. Os exemplos incluem ressonância magnética de difusão , ressonância magnética funcional e outros.
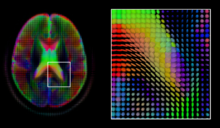
Ressonância Magnética de Difusão
A ressonância magnética por difusão é uma modalidade de imagem de ressonância magnética estrutural que permite a medição do processo de difusão de moléculas. A difusão é medida aplicando um pulso de gradiente a um campo magnético ao longo de uma direção particular. Em uma aquisição típica, um conjunto de direções de gradiente uniformemente distribuídas é usado para criar um conjunto de volumes de difusão ponderada. Além disso, um volume não ponderado é adquirido sob o mesmo campo magnético sem a aplicação de um pulso gradiente. Como cada aquisição está associada a vários volumes, a difusão MRI criou uma variedade de desafios únicos na computação de imagens médicas.
Na medicina, existem dois objetivos computacionais principais na ressonância magnética de difusão :
- Estimativa de propriedades locais do tecido, como difusividade;
- Estimativa de direções locais e vias globais de difusão.
O tensor de difusão , uma matriz simétrica positiva-definida 3 × 3 , oferece uma solução direta para ambos os objetivos. É proporcional à matriz de covariância de um perfil de difusão local normalmente distribuído e, portanto, o autovetor dominante dessa matriz é a direção principal da difusão local. Devido à simplicidade deste modelo, uma estimativa de máxima verossimilhança do tensor de difusão pode ser encontrada simplesmente resolvendo um sistema de equações lineares em cada local de forma independente. No entanto, como se presume que o volume contém fibras de tecido contíguas, pode ser preferível estimar o volume dos tensores de difusão em sua totalidade, impondo condições de regularidade no campo subjacente de tensores. Os valores escalares podem ser extraídos do tensor de difusão, como a anisotropia fracionada , difusividades média, axial e radial, que medem indiretamente propriedades do tecido como a desmielinização de fibras axonais ou a presença de edema. Métodos de computação de imagem escalar padrão, como registro e segmentação, podem ser aplicados diretamente a volumes de tais valores escalares. No entanto, para explorar totalmente as informações no tensor de difusão, esses métodos foram adaptados para contabilizar os volumes com valores de tensor ao realizar o registro e a segmentação.
Dada a direção principal de difusão em cada local do volume, é possível estimar as vias globais de difusão por meio de um processo conhecido como tractografia . No entanto, devido à resolução relativamente baixa da difusão por ressonância magnética , muitas dessas vias podem se cruzar, se beijar ou se espalhar em um único local. Nesta situação, a única direção principal do tensor de difusão não é um modelo apropriado para a distribuição de difusão local. A solução mais comum para este problema é estimar múltiplas direções de difusão local usando modelos mais complexos. Isso inclui misturas de tensores de difusão, imagem Q-ball, imagem de espectro de difusão e funções de distribuição de orientação de fibra, que normalmente requerem aquisição HARDI com um grande número de direções de gradiente. Tal como acontece com o tensor de difusão, os volumes avaliados com esses modelos complexos requerem tratamento especial ao aplicar métodos de computação de imagem, como registro e segmentação.
Ressonância magnética funcional
A ressonância magnética funcional (fMRI) é uma modalidade de imagem médica que mede indiretamente a atividade neural por meio da observação da hemodinâmica local ou do sinal dependente do nível de oxigênio no sangue (BOLD). Os dados de fMRI oferecem uma variedade de percepções e podem ser divididos em duas categorias:
- O fMRI relacionado à tarefa é adquirido quando o sujeito está realizando uma sequência de condições experimentais cronometradas. Em experimentos de design de bloco, as condições estão presentes por curtos períodos de tempo (por exemplo, 10 segundos) e são alternadas com períodos de descanso. Os experimentos relacionados a eventos contam com uma sequência aleatória de estímulos e usam um único ponto no tempo para denotar cada condição. A abordagem padrão para analisar fMRI relacionada à tarefa é o modelo linear geral (GLM)
- O estado de repouso fMRI é adquirido na ausência de qualquer tarefa experimental. Normalmente, o objetivo é estudar a estrutura de rede intrínseca do cérebro. As observações feitas durante o repouso também foram vinculadas a processos cognitivos específicos, como codificação ou reflexão. A maioria dos estudos de fMRI em estado de repouso concentra-se nas flutuações de baixa frequência do sinal de fMRI (LF-BOLD). As descobertas seminais incluem a rede padrão , uma parcela cortical abrangente e a vinculação das características da rede a parâmetros comportamentais.
Existe um rico conjunto de metodologia usada para analisar dados de neuroimagem funcional e, muitas vezes, não há consenso sobre o melhor método. Em vez disso, os pesquisadores abordam cada problema de forma independente e selecionam um modelo / algoritmo adequado. Nesse contexto, há um intercâmbio relativamente ativo entre as comunidades de neurociência , biologia computacional , estatística e aprendizado de máquina . Abordagens proeminentes incluem
- Abordagens univariadas massivas que investigam voxels individuais nos dados de imagem para uma relação com a condição do experimento. A abordagem principal é o modelo linear geral (GLM)
- Abordagens multivariadas e baseadas em classificadores , frequentemente chamadas de análise de padrão multi voxel ou análise de padrão multivariada sondam os dados para respostas globais e potencialmente distribuídas a uma condição experimental. As primeiras abordagens usavam máquinas de vetores de suporte (SVM) para estudar as respostas aos estímulos visuais. Recentemente, algoritmos alternativos de reconhecimento de padrões foram explorados, como contraste gini baseado em floresta aleatória ou regressão esparsa e aprendizado de dicionário
- A análise de conectividade funcional estuda a estrutura de rede intrínseca do cérebro, incluindo as interações entre as regiões. A maioria desses estudos concentra-se nos dados do estado de repouso para dividir o cérebro ou encontrar correlações com medidas comportamentais. Os dados específicos da tarefa podem ser usados para estudar as relações causais entre as regiões do cérebro (por exemplo, mapeamento causal dinâmico (DCM)).
Ao trabalhar com grandes coortes de sujeitos, a normalização (registro) de sujeitos individuais em um referencial comum é crucial. Um corpo de trabalho e ferramentas existem para realizar a normalização com base na anatomia ( FSL , FreeSurfer , SPM ). O alinhamento que leva em conta a variabilidade espacial entre os assuntos é uma linha de trabalho mais recente. Os exemplos são o alinhamento do córtex com base na correlação de sinal de fMRI, o alinhamento com base na estrutura de conectividade funcional global em dados de tarefa ou de estado de repouso e o alinhamento com base em perfis de ativação específicos de estímulos de voxels individuais.
Programas
O software para computação de imagens médicas é uma combinação complexa de sistemas que fornecem IO, visualização e interação, interface de usuário, gerenciamento de dados e computação. Normalmente, as arquiteturas de sistema são dispostas em camadas para atender desenvolvedores de algoritmos, desenvolvedores de aplicativos e usuários. As camadas inferiores são geralmente bibliotecas e / ou kits de ferramentas que fornecem recursos computacionais básicos; enquanto as camadas superiores são aplicativos especializados que tratam de problemas médicos, doenças ou sistemas corporais específicos.
Notas Adicionais
A computação de imagens médicas também está relacionada ao campo da visão computacional . Uma sociedade internacional, a Sociedade MICCAI representa o campo e organiza uma conferência anual e workshops associados. Os procedimentos desta conferência foram publicados pela Springer na série Lecture Notes in Computer Science. Em 2000, N. Ayache e J. Duncan revisaram o estado do campo.
Veja também
Referências
Revistas sobre computação de imagens médicas
- Análise de imagens médicas (MedIA) ; também o jornal oficial da The MICCAI Society , que organiza a Conferência Anual MICCAI, uma conferência importante para a computação de imagens médicas
- IEEE Transactions on Medical Imaging (IEEE TMI)
- Física médica
- Journal of Digital Imaging (JDI) ; o jornal oficial da Society of Imaging Informatics
- Imagens e gráficos médicos computadorizados
- Journal of Computer Aided Radiology and Surgery
- BMC Medical Imaging
Além disso, os periódicos a seguir publicam ocasionalmente artigos que descrevem métodos e aplicações clínicas específicas de computação de imagens médicas ou computação de imagens médicas de modalidade específica