Metilação de histona - Histone methylation
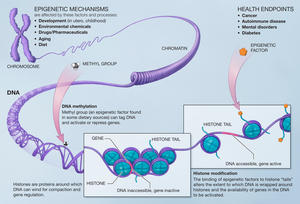
A metilação das histonas é um processo pelo qual os grupos metil são transferidos para os aminoácidos das proteínas histonas que formam os nucleossomos , que a dupla hélice do DNA envolve para formar os cromossomos . A metilação das histonas pode aumentar ou diminuir a transcrição dos genes, dependendo de quais aminoácidos nas histonas são metilados e de quantos grupos metil estão ligados. Os eventos de metilação que enfraquecem as atrações químicas entre as caudas das histonas e o DNA aumentam a transcrição porque permitem que o DNA se desenrole dos nucleossomos para que as proteínas do fator de transcrição e a RNA polimerase possam acessar o DNA. Este processo é crítico para a regulação da expressão gênica que permite que diferentes células expressem diferentes genes.
Função
A metilação das histonas, como mecanismo de modificação da estrutura da cromatina , está associada à estimulação de vias neurais conhecidas por serem importantes para a formação de memórias e aprendizado de longo prazo . Modelos animais mostraram que a metilação e outros mecanismos de regulação epigenética estão associados a condições de envelhecimento, doenças neurodegenerativas e deficiência intelectual ( síndrome de Rubinstein-Taybi , deficiência intelectual ligada ao X ). A desregulação de H3K4, H3K27 e H4K20 está associada a cânceres . Essa modificação altera as propriedades do nucleossomo e afeta suas interações com outras proteínas, principalmente no que diz respeito aos processos de transcrição gênica.
- A metilação das histonas pode estar associada à repressão transcricional ou ativação . Por exemplo, a trimetilação da histona H3 na lisina 4 ( H3K4me3 ) é uma marca ativa para a transcrição e é regulada positivamente no hipocampo uma hora após o condicionamento contextual do medo em ratos. No entanto, a dimetilação da histona H3 na lisina 9 ( H3K9me2 ), um sinal para o silenciamento transcricional, é aumentada após a exposição ao condicionamento do medo ou a um novo ambiente sozinho.
- A metilação de alguns resíduos de lisina (K) e arginina (R) de histonas resulta na ativação transcricional. Os exemplos incluem metilação de lisina 4 de histona 3 ( H3K4me1 ) e resíduos de arginina (R) em H3 e H4 .
- A adição de grupos metil às histonas por histona metiltransferases pode ativar ou ainda reprimir a transcrição, dependendo do aminoácido sendo metilado e da presença de outros grupos metil ou acetil na vizinhança.
Mecanismo
A unidade fundamental da cromatina , chamada de nucleossomo , contém DNA enrolado em torno de um octâmero de proteína . Este octâmero consiste em duas cópias de cada uma das quatro proteínas histonas: H2A , H2B , H3 e H4 . Cada uma dessas proteínas tem uma extensão de cauda, e essas caudas são os alvos da modificação do nucleossomo por metilação. A ativação ou inativação do DNA é amplamente dependente do resíduo específico da cauda metilado e de seu grau de metilação. As histonas podem ser metiladas em resíduos de lisina (K) e arginina (R) apenas, mas a metilação é mais comumente observada em resíduos de lisina das caudas de histona H3 e H4. A extremidade da cauda mais distante do núcleo do nucleossomo é o terminal N (os resíduos são numerados começando nesta extremidade). Os locais comuns de metilação associados à ativação do gene incluem H3K4, H3K48 e H3K79. Os locais comuns para a inativação de genes incluem H3K9 e H3K27. Estudos desses locais descobriram que a metilação das caudas das histonas em diferentes resíduos servem como marcadores para o recrutamento de várias proteínas ou complexos de proteínas que servem para regular a ativação ou inativação da cromatina.
Os resíduos de lisina e arginina contêm grupos amino, que conferem características básicas e hidrofóbicas. A lisina pode ser mono-, di- ou trimetilada com um grupo metil substituindo cada hidrogênio de seu grupo NH3 +. Com um grupo NH2 e NH2 + livre, a arginina pode ser mono- ou dimetilada. Esta dimetilação pode ocorrer assimetricamente no grupo NH2 ou simetricamente com uma metilação em cada grupo. Cada adição de um grupo metil em cada resíduo requer um conjunto específico de enzimas proteicas com vários substratos e cofatores. Geralmente, a metilação de um resíduo de arginina requer um complexo incluindo a proteína arginina metiltransferase (PRMT), enquanto a lisina requer uma histona metiltransferase específica (HMT), geralmente contendo um domínio SET evolutivamente conservado.
Diferentes graus de metilação do resíduo podem conferir diferentes funções, como exemplificado na metilação do resíduo H4K20 comumente estudado. O H4K20 monometilado ( H4K20me 1) está envolvido na compactação da cromatina e, portanto, na repressão transcricional. No entanto, o H4K20me2 é vital no reparo de DNA danificado. Quando dimetilado, o resíduo fornece uma plataforma para a ligação da proteína 53BP1 envolvida no reparo de quebras de DNA de fita dupla por união de extremidade não homóloga. Observou-se que H4K20me3 está concentrado em heterocromatina e reduções nesta trimetilação são observadas na progressão do câncer. Portanto, o H4K20me3 desempenha um papel adicional na repressão da cromatina. O reparo de quebras de fita dupla de DNA na cromatina também ocorre por recombinação homóloga e também envolve metilação de histonas ( H3K9me3 ) para facilitar o acesso das enzimas de reparo aos locais de dano.
Histona metiltransferase
O genoma está fortemente condensado em cromatina, que precisa ser afrouxada para que a transcrição ocorra. Para interromper a transcrição de um gene, o DNA deve ser enrolado com mais firmeza. Isso pode ser feito modificando as histonas em certos locais por metilação. As histonas metiltransferases são enzimas que transferem grupos metila de S-adenosil metionina (SAM) para os resíduos de lisina ou arginina das histonas H3 e H4. Existem casos de domínios globulares centrais de histonas sendo metilados também.
As metiltransferases histonas são específicas para lisina ou arginina. As transferases específicas da lisina são ainda divididas em se elas têm ou não um domínio SET ou um domínio não SET. Esses domínios especificam exatamente como a enzima catalisa a transferência do metil do SAM para a proteína de transferência e posteriormente para o resíduo de histona. As metiltransferases podem adicionar 1-3 metilos nos resíduos alvo.
Essas metilas que são adicionadas às histonas agem para regular a transcrição, bloqueando ou encorajando o acesso do DNA aos fatores de transcrição. Desta forma, a integridade do genoma e a herança epigenética dos genes estão sob o controle das ações das metiltransferases histonas. A metilação das histonas é a chave para distinguir a integridade do genoma e os genes que são expressos pelas células, dando às células suas identidades.
As histonas metiladas podem reprimir ou ativar a transcrição. Por exemplo, enquanto H3K4me2 , H3K4me3 e H3K79me3 estão geralmente associados à atividade transcricional, enquanto H3K9me2 , H3K9me3 , H3K27me2 , H3K27me3 e H4K20me3 estão associados à repressão transcricional.
Epigenética
As modificações feitas na histona têm um efeito nos genes que são expressos em uma célula e é o caso quando metilas são adicionadas aos resíduos de histona pelas metiltransferases de histona. A metilação das histonas desempenha um papel importante na montagem do mecanismo de heterocromatina e na manutenção das fronteiras gênicas entre os genes que são transcritos e os que não são. Essas mudanças são transmitidas à progênie e podem ser afetadas pelo ambiente ao qual as células estão sujeitas. As alterações epigenéticas são reversíveis, o que significa que podem ser alvos de terapia.
As atividades das histonas metiltransferases são compensadas pela atividade das histonas desmetilases. Isso permite ligar ou desligar a transcrição, revertendo as modificações pré-existentes. É necessário que as atividades das histonas metiltrasnsferases e histonas demetilases sejam reguladas rigidamente. A desregulação de qualquer um dos dois pode levar à expressão gênica que leva ao aumento da suscetibilidade à doença. Muitos cânceres surgem dos efeitos epigenéticos inadequados da metilação desregulada. No entanto, como esses processos às vezes são reversíveis, há interesse em utilizar suas atividades em conjunto com terapias anticâncer.
Na inativação do cromossomo X
Nos organismos femininos, um espermatozóide contendo um cromossomo X fertiliza o óvulo, dando ao embrião duas cópias do cromossomo X. As mulheres, no entanto, inicialmente não precisam de ambas as cópias do cromossomo X, pois isso apenas dobraria a quantidade de produtos proteicos transcritos, conforme mostrado pela hipótese de compensação de dosagem. O cromossomo X paterno é rapidamente inativado durante as primeiras divisões. Este cromossomo X inativo (Xi) é embalado em uma forma incrivelmente compacta de cromatina chamada heterocromatina . Esse empacotamento ocorre devido à metilação dos diferentes resíduos de lisina que ajudam a formar diferentes histonas. Em humanos, a inativação do X é um processo aleatório, que é mediado pelo RNA não codificador XIST.
Embora a metilação de resíduos de lisina ocorra em muitas histonas diferentes, a mais característica de Xi ocorre na nona lisina da terceira histona (H3K9). Enquanto uma única metilação desta região permite que os genes ligados permaneçam transcricionalmente ativos, na heterocromatina esse resíduo de lisina é frequentemente metilado duas ou três vezes, H3K9me2 ou H3K9me3 , respectivamente, para garantir que o DNA ligado seja inativo. Pesquisas mais recentes mostraram que H3K27me3 e H4K20me1 também são comuns em embriões em estágio inicial. Outras marcações de metilação associadas a áreas transcricionalmente ativas do DNA, H3K4me2 e H3K4me3, estão ausentes no cromossomo Xi, juntamente com muitas marcações de acetilação. Embora fosse sabido que certas marcações de metilação da histona Xi permaneceram relativamente constantes entre as espécies, foi recentemente descoberto que diferentes organismos e até mesmo células diferentes dentro de um único organismo podem ter marcações diferentes para sua inativação de X. Por meio da metilação das histonas, há impressão genética , de modo que o mesmo homólogo X permanece inativado por meio de replicações cromossômicas e divisões celulares.
Mutações
Devido ao fato de que a metilação das histonas regula muitos dos genes que são transcritos, mesmo pequenas mudanças nos padrões de metilação podem ter efeitos terríveis no organismo. Mutações que ocorrem para aumentar e diminuir a metilação têm grandes mudanças na regulação do gene, enquanto mutações em enzimas como metiltransferase e desmetiltransferase podem alterar completamente quais proteínas são transcritas em uma determinada célula. A metilação excessiva de um cromossomo pode fazer com que certos genes que são necessários para o funcionamento normal da célula se tornem inativados. Em uma certa cepa de levedura, Saccharomyces cerevisiae, uma mutação que faz com que três resíduos de lisina na terceira histona, H3K4, H3K36 e H3K79, se tornem metilados, causa um atraso no ciclo celular mitótico, pois muitos genes necessários para essa progressão são inativados. Essa mutação extrema leva à morte do organismo. Foi descoberto que a deleção de genes que irão eventualmente permitir a produção de histona metiltransferase permite que este organismo viva, pois seus resíduos de lisina não são metilados.
Nos últimos anos, chamou a atenção dos pesquisadores que muitos tipos de câncer são causados em grande parte devido a fatores epigenéticos. O câncer pode ser causado de várias maneiras devido à metilação diferencial das histonas. Desde a descoberta dos oncogenes , bem como dos genes supressores de tumor , sabe-se que um grande fator de causar e reprimir o câncer está em nosso próprio genoma. Se as áreas ao redor dos oncogenes se tornarem não metiladas, esses genes causadores de câncer têm o potencial de ser transcritos a uma taxa alarmante. O oposto disso é a metilação dos genes supressores de tumor. Nos casos em que as áreas ao redor desses genes eram altamente metiladas, o gene supressor de tumor não estava ativo e, portanto, o câncer era mais provável de ocorrer. Essas mudanças no padrão de metilação geralmente são devidas a mutações na metiltransferase e na desmetiltransferase. Outros tipos de mutações em proteínas, como isocitrato desidrogenase 1 (IDH1) e isocitrato desidrogenase 2 (IDH2), podem causar a inativação da histona demetiltransferase que, por sua vez, pode levar a uma variedade de cânceres, gliomas e leucemias, dependendo de quais células a mutação ocorre.
O metabolismo de um carbono modifica a metilação da histona
No metabolismo de um carbono, os aminoácidos glicina e serina são convertidos por meio dos ciclos de folato e metionina em precursores de nucleotídeos e SAM. Vários nutrientes alimentam o metabolismo de um carbono, incluindo glicose, serina, glicina e treonina. Altos níveis do doador de metila SAM influenciam a metilação das histonas, o que pode explicar como altos níveis de SAM evitam a transformação maligna.
Veja também
- Código histone
- Acetilação e desacetilação de histonas
- Histona metiltransferase
- Metilação
- Metilisina
- Impressão Genética
Referências
Leitura adicional
- Orouji, Elias & Utikal, Jochen. (2018). Combate ao melanoma maligno epigeneticamente: metilação de histona lisina. Clinical Epigenetics 2018 10: 145 https://clinicalepigeneticsjournal.biomedcentral.com/articles/10.1186/s13148-018-0583-z
- Gozani, O., & Shi, Y. (2014). Metilação de histonas na sinalização da cromatina. In: Fundamentals of Chromatin (pp. 213–256). Springer New York. doi : 10.1007 / 978-1-4614-8624-4_5
- Shi, Yang; Whetstine, Johnathan R. (12 de janeiro de 2007). "Dynamic Regulation of Histone Lysine Methylation by Demethylases" . Molecular Cell . 25 (1): 1–14. doi : 10.1016 / j.molcel.2006.12.010 . PMID 17218267 .