BLAT (bioinformática) - BLAT (bioinformatics)
| Desenvolvedor (s) | Jim Kent , UCSC |
|---|---|
| Repositório |
|
| Tipo | ferramenta de bioinformática |
| Licença | gratuito para uso não comercial , fonte disponível |
| Local na rede Internet | genoma |
BLAT ( BLAST -como ferramenta de alinhamento) é um alinhamento de sequência de pares algoritmo que foi desenvolvido por Jim Kent na Universidade da Califórnia, Santa Cruz (UCSC) no início dos anos 2000 para ajudar na montagem e anotação do genoma humano . Foi concebido principalmente para diminuir o tempo necessário para alinhar milhões de rato genómico leituras e expressa marcadores de sequência contra a sequência do genoma humano. As ferramentas de alinhamento do tempo não foram capazes de realizar estas operações de uma maneira que permita uma actualização regular da montagem do genoma humano. Em comparação com ferramentas pré-existentes, BLAT foi ~ 500 vezes mais rápido com a realização de mRNA / DNA alinhamentos e ~ 50 vezes mais rápido com proteína alinhamentos / proteína.
Conteúdo
visão global
BLAT é um de vários algoritmos desenvolvidos para a análise e comparação de sequências biológicas, tais como DNA, RNA e proteínas, com o objectivo principal de inferir de homologia , de modo a descobrir a função biológica de sequências genómicas. Não é garantido para encontrar o alinhamento matematicamente ideal entre duas sequências como o clássico Needleman-Wunsch e Smith-Waterman programação dinâmica algoritmos fazer; em vez disso, primeiro tenta detectar rapidamente sequências curtas que são mais susceptíveis de serem homólogas, e, em seguida, se alinha e se estende ainda mais as regiões homólogas. É semelhante à heurística família BLAST de algoritmos, mas cada ferramenta tem tentado lidar com o problema de alinhamento de sequências biológicas de uma maneira oportuna e eficiente, tentando diferentes técnicas algorítmicas.
Usos de BLAT
BLAT pode ser utilizada para alinhar sequcias de ADN, bem como proteínas e nucleótidos traduzida sequências (de ARNm ou de ADN). Ele é projetado para funcionar melhor em seqüências com grande semelhança. A pesquisa DNA é mais eficaz para primatas ea busca proteína é eficaz para vertebrados terrestres. Além disso, a proteína ou consultas sequência traduzida são mais eficazes para a identificação de jogos distantes e para análise de cruzamento de espécies de consultas de sequência de ADN. Os usos típicos de BLAT incluem o seguinte:
- Alinhamento de múltiplas sequências de ARNm para uma montagem do genoma, a fim de inferir as suas coordenadas genómicos;
- Alinhamento de uma sequência de proteína ou de ARNm a partir de uma espécie para uma base de dados de sequência a partir de uma outra espécie para determinar a homologia. Desde que as duas espécies não são demasiado divergentes, o alinhamento de espécies cruzadas é geralmente eficaz com BLAT. Isto é possível porque BLAT não exige fósforos perfeitos, mas sim aceita descasamentos de alinhamentos;
- BLAT podem ser utilizados para os alinhamentos de duas sequências de proteína. No entanto, não é a ferramenta de escolha para estes tipos de alinhamentos. BLASTP, a proteína padrão BLAST ferramenta, é mais eficiente em alinhamentos proteína-proteína;
- A determinação da distribuição de regiões intrónicas exicas e de um gene;
- Detecção de membros da família de genes de uma consulta gene específico;
- Indicação da sequência de codificação da proteína de um gene específico.
BLAT foi concebido para encontrar correspondências entre as sequências de comprimento pelo menos 40 bases que partilham ≥95% de identidade de nucleótidos ou ≥80% de identidade de proteína traduzida.
Processo
BLAT é utilizado para encontrar as regiões em uma base de dados genica alvo, que são semelhantes a uma sequência de consulta sob exame. O processo geral seguido por algorítmica BLAT é semelhante ao BLAST 's em que ele procura primeiro por segmentos curtos nas sequências de bancos de dados e de consulta, que têm um certo número de elementos correspondentes. Estas sementes de alinhamento são então estendidas em ambas as direcções das sequências, a fim de formar pares de alta pontuação. No entanto, BLAT utiliza uma abordagem diferente de indexação BLAST, o que lhe permite digitalizar rapidamente grandes bancos de dados genómicos e de proteína para semelhanças com uma sequcia de pesquisa. Ela faz isso por manter uma lista indexada ( tabela hash ) da base de dados na memória alvo, o que reduz significativamente o tempo requerido para a comparação das sequências de consulta com o banco de dados de alvo. Este índice é construído tomando as coordenadas de todos os não-sobrepostas k-mers (palavras com letras k) no banco de dados de alvo, com excepção para a k-mers altamente repetidos. BLAT então constrói uma lista de todos os k-mers sobrepostos da sequência de consulta e pesquisas para estes na base de dados de alvo, construindo-se uma lista de resultados onde existem partidas entre as sequências (Figura 1 ilustra este processo).
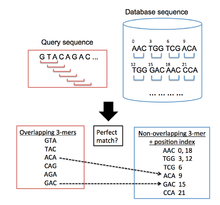
estágio de pesquisa
Há três estratégias diferentes usados, a fim de procurar regiões homólogas candidatos:
- O primeiro método requer fósforos perfeitos simples entre as seqüências de consulta e de banco de dados ou seja, as duas palavras k-mer são exatamente os mesmos. Esta abordagem não é considerado o mais prático. Isto porque um pequeno tamanho k-mer é necessário a fim de alcançar níveis elevados de sensibilidade, mas isto aumenta o número de visitas de falsos positivos, aumentando assim a quantidade de tempo gasto na fase de alinhamento do algoritmo.
- O segundo método permite, pelo menos, uma incompatibilidade entre as duas palavras k-mer. Isso diminui a quantidade de falsos positivos, permitindo tamanhos de k-mer maiores que são menos computacionalmente dispendioso de manusear do que as produzidas a partir do método anterior. Este método é muito eficaz na identificação de pequenas regiões homólogas.
- O terceiro método requer múltiplas combinações perfeitas que estão em estreita proximidade um do outro. Como mostra Kent, esta é uma técnica muito eficaz capaz de levar em consideração as pequenas inserções e deleções dentro das regiões homólogas.
Ao alinhar nucleótidos, BLAT usa o terceiro método que requer duas correspondências de palavras perfeitas de tamanho 11 (11-meros). Ao alinhar proteínas, a versão BLAT determina a metodologia de pesquisa utilizada: quando a versão cliente / servidor é usado, pesquisas CLAT por três perfeitos jogos 4-meros; quando a versão stand-alone é usado, pesquisas CLAT para um único perfeito 5-mer entre as seqüências de consulta e banco de dados.
BLAST contra BLAT
Algumas das diferenças entre BLAT e BLAST estão descritas a seguir:
- índices de bases de dados do genoma CLAT / proteína, retém o índice em memória, e em seguida, examina a sequência de consulta para partidas. BLAST, por outro lado, cria um índice das sequências de consulta e pesquisas na base de dados para os jogos. Uma variante BLAST chamado índices megablast 4 bases de dados para acelerar alinhamentos.
- BLAT pode estender-se em várias combinações perfeitas e quase perfeitas (padrão é 2 partidas perfeitas de comprimento 11, para pesquisas de nucleidos e 3 partidas perfeitas de comprimento 4 para pesquisas de proteína), enquanto BLAST estende-se apenas quando uma ou duas partidas ocorrem próximas umas das outras.
- BLAT requer seqüências de consulta em formato FASTA , enquanto BLAST aceita ambas as sequências e consultas formatado-FASTA pelo número de acesso .
- BLAT conecta cada homólogo área entre duas sequências em um único alinhamento maior, em contraste com BLAST que retorna cada área homóloga como um alinhamento local separado. O resultado de BLAST é uma lista de exs com cada alinhamento estendendo-se apenas para além da extremidade do exão. BLAT, no entanto, coloca correctamente cada base do ARNm para o genoma, utilizando cada base apenas uma vez e pode ser usado para identificar intrão limites -exon (ou seja, locais de splicing ).
- BLAT é menos sensível do que BLAST.
uso do programa
BLAT pode ser usado tanto como um programa cliente-servidor baseada na Web ou como um programa stand-alone.
Cliente-servidor
O aplicativo baseado na web de BLAT pode ser acessado a partir do UCSC Genome Bioinformatics Site. Construir o índice é um procedimento relativamente lento. Portanto, cada montagem do genoma utilizado pelo BLAT baseado na web está associado com um servidor BLAT, a fim de ter um índice de pré-calculado disponível para os alinhamentos. Esses servidores CLAT baseados na web manter o índice na memória para os usuários a entrada de suas seqüências de consulta.
Uma vez que a sequência de consulta é enviado / colado no campo de pesquisa, o utilizador pode seleccionar vários parâmetros, tais como o que genoma da espécie ao alvo (existem actualmente mais de 50 espécies disponíveis) e a versão a montagem desse genoma (por exemplo, o genoma humano tem quatro conjuntos a partir de escolha), o tipo de consulta (ou seja, se a sequência de ADN refere-se a, etc. definições de saída de proteína) e (isto é, como classificar e visualizar a saída). O usuário pode então executar a busca por qualquer submeter a consulta ou usando a busca BLAT "Estou com sorte".
Bhagwat et ai. fornecer passo a passo protocolos de como usar BLAT para:
- Mapear uma sequcia de ARNm / ADNc, para uma sequência genómica;
- Mapear uma sequcia de protea para o genoma;
- Realizar pesquisas de homologia.
Entrada
BLAT pode lidar com seqüências de banco de dados longos, no entanto, é mais eficaz com seqüências de consulta curtas do que as sequências de consulta longas. Kent recomenda um comprimento máximo de consulta de 200.000 bases. O navegador UCSC limita sequências de consulta para menos do que 25.000 letras (isto é, nucleótidos ) para DNA pesquisas e menos do que 10.000 letras (isto é, aminoácidos ) para proteína e pesquisas sequência traduzida.
O BLAT Pesquisa do Genoma disponível no site da UCSC aceita seqüências de consulta como texto (recortado e colado na caixa de consulta) ou enviados como arquivos de texto. O BLAT Pesquisa Genoma pode aceitar várias sequências do mesmo tipo de uma só vez, até um máximo de 25. Para várias sequências, o número total de nucleótidos não deve exceder 50.000 para pesquisas de ADN ou 25.000 cartas para a proteína ou pesquisas sequência traduzida. Um exemplo de uma base de dados procura alvo com uma sequência de pesquisa de ADN é mostrada na Figura 2.
Saída
Uma busca BLAT retorna uma lista de resultados que são ordenados em ordem com base na pontuação decrescente. O seguinte informação é devolvido: a pontuação do alinhamento, a região da sequência de consulta que corresponde à sequência da base de dados, o tamanho da sequência de consulta, o nível de identidade em percentagem do alinhamento e o cromossoma e da posição que a sequcia de pesquisa mapas para. Bhagwat et ai. descrever como o BLAT "Score" e medidas de "identidade" são calculados.
Para cada resultado da pesquisa, o usuário é fornecido com um link para o navegador UCSC Genome para que eles possam visualizar o alinhamento no cromossomo. Este um grande benefício do BLAT baseado na web sobre o BLAT stand-alone. O utilizador é capaz de obter informação biológica associada com o alinhamento, tal como a informação sobre o gene para o qual a consulta pode corresponder. O utilizador é também proporcionado com uma ligação para visualizar o alinhamento da sequcia problema com a montagem do genoma. As partidas entre o conjunto de consultas e genoma são azuis e os limites dos alinhamentos são de cor mais clara. Estes limites de exão indicam locais de splicing. O resultado da pesquisa "Estou com sorte" retorna o alinhamento de pontuação mais alta para a primeira seqüência de consulta com base na opção de classificação de saída selecionado pelo usuário.
Estar sozinho
BLAT Stand-alone é mais adequado para execuções em batch, e mais eficiente do que o BLAT baseado na Web. É mais eficiente porque é capaz de armazenar o genoma na memória, ao contrário do aplicativo baseado na web que armazena apenas o índice na memória.
Licença
Tanto a fonte e binários pré-compilados de BLAT estão disponíveis gratuitamente para uso acadêmico e pessoal. Licença comercial de BLAT stand-alone é distribuído por Kent Informática, Inc.
Veja também
- BLAST Básico Local Alignment Search Tool
- software de alinhamento de sequências

