Efetora semelhante a ativador de transcrição - Transcription activator-like effector
| |||||||
| Identificadores | |||||||
|---|---|---|---|---|---|---|---|
| Organismo | |||||||
| Símbolo | pthXo1 | ||||||
| RefSeq (Prot) | WP_041182630.1 | ||||||
| UniProt | B2SU53 | ||||||
| Outros dados | |||||||
| Cromossoma | Genômico: 1,65 - 1,65 Mb | ||||||
| |||||||
TAL ( activador de tipo transcrição ) efectores (muitas vezes referida como contos , mas não deve ser confundido com o t rês um mino ácido l oop e xtension classe homeobox de proteínas) são proteínas segregadas por alguns β- e γ-Proteobacteria . A maioria deles são Xanthomonads . As bactérias Xanthomonas patogênicas de plantas são especialmente conhecidas por TALEs, produzidas por meio de seu sistema de secreção tipo III . Essas proteínas podem se ligar a sequências promotoras na planta hospedeira e ativar a expressão de genes de plantas que auxiliam na infecção bacteriana. Eles reconhecem sequências de DNA de plantas por meio de um domínio de repetição central que consiste em um número variável de ~ 34 repetições de aminoácidos. Parece haver uma correspondência de um para um entre a identidade de dois aminoácidos críticos em cada repetição e cada base de DNA na sequência alvo. Essas proteínas são interessantes para os pesquisadores tanto por seu papel na doença de espécies importantes de plantações quanto pela relativa facilidade de redirecioná-las para se ligarem a novas sequências de DNA. Proteínas semelhantes podem ser encontradas na bactéria patogênica Ralstonia solanacearum e Burkholderia rhizoxinica , bem como em microrganismos marinhos ainda não identificados. O termo TALE-like é usado para se referir à família de proteínas putativa que abrange os TALEs e essas proteínas relacionadas.
Função na patogênese vegetal
Xanthomonas
Xanthomonas são bactérias Gram-negativas que podem infectar uma ampla variedade de espécies de plantas, incluindo pimenta / capsicum, arroz, frutas cítricas, algodão, tomate e soja. Alguns tipos de Xanthomonas causam manchas foliares localizadas ou estrias foliares, enquanto outros se espalham sistemicamente e causam podridão negra ou doença da podridão-das-folhas. Eles injetam várias proteínas efetoras, incluindo efetores TAL, na planta por meio de seu sistema de secreção tipo III . Os efetores TAL têm vários motivos normalmente associados a eucariotos, incluindo múltiplos sinais de localização nuclear e um domínio de ativação ácida. Quando injetadas em plantas, essas proteínas podem entrar no núcleo da célula vegetal, ligar-se a sequências promotoras de plantas e ativar a transcrição de genes de plantas que auxiliam na infecção bacteriana. As plantas desenvolveram um mecanismo de defesa contra os efetores do tipo III que inclui os genes R (resistência) acionados por esses efetores. Alguns desses genes R parecem ter evoluído para conter locais de ligação efetores de TAL semelhantes ao local no gene alvo pretendido. Essa competição entre as bactérias patogênicas e a planta hospedeira foi considerada responsável pela natureza aparentemente maleável do domínio de ligação ao DNA efetor de TAL.
| X. espécies | colheita de vítima |
|---|---|
| X. campestris pv. musacerum , X. vasicola pv. musacero | banana e ensete |
| X. axonopodis pv. phaseoli , X. axonopodis pv. glicinas | feijão comum e soja |
| X. axonopodis pv. vignicola | feijão frade |
| X. campestris pv. campestris , X. campestris pv. armoracieae | todos Brassica s |
| X. axonopodis pv. manihotis | mandioca |
| X. translucens pv. translúcidos | cevada , centeio , trigo , triticale |
| X. axonopodis pv. citri , X. citri , X. citri pv. aurantifolii | citrino |
| X. campestris pv. Malvacearum | algodão |
| X. euvesicatoria , X. gardneri | pimenta / pimentão |
| X. oryzae pv. Oryzae | arroz |
| X. arboricola pv. pruni | ameixa , pêssego , nectarina |
| X. perforans , X. campestris pv. vesicatória | tomate |
| X. campestris pv. mangiferaindicae | manga |
| X. arboricola pv. Juglandis | noz |
Não Xanthomonas
R. solanacearum , B. rhizoxinica e doença do sangue da banana (uma bactéria ainda não identificada definitivamente, no grupo de espécies de R. solanacearum ).
Reconhecimento de DNA
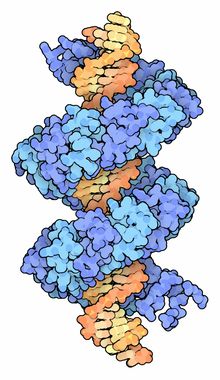
A característica mais distinta dos efetores TAL é um domínio de repetição central contendo entre 1,5 e 33,5 repetições que geralmente têm 34 resíduos de comprimento (a repetição C-terminal é geralmente mais curta e referida como uma "meia repetição"). Uma sequência de repetição típica é LTPEQVVAIAS HD GGKQALETVQRLLPVLCQAHG , mas os resíduos nas posições 12ª e 13ª são hipervariáveis (estes dois aminoácidos são também conhecidos como resíduo direto variável de repetição ou RVD). Há uma relação simples entre a identidade desses dois resíduos em repetições sequenciais e bases de DNA sequenciais no local alvo do efetor TAL. A estrutura cristalina de um efetor TAL ligado ao DNA indica que cada repetição compreende duas hélices alfa e uma alça contendo RVD curta, onde o segundo resíduo do RVD faz contatos de DNA específicos para a sequência, enquanto o primeiro resíduo do RVD estabiliza o que contém RVD ciclo. Os locais alvo dos efetores TAL também tendem a incluir uma timina flanqueando a base 5 'direcionada pela primeira repetição; isto parece ser devido a um contato entre este T e um triptofano conservado na região N-terminal do domínio de repetição central. No entanto, essa posição "zero" nem sempre contém uma timina, pois alguns andaimes são mais permissivos.
O código TAL-DNA foi quebrado por dois grupos separados em 2010. O primeiro grupo, liderado por Adam Bogdanove , quebrou esse código computacionalmente ao procurar padrões em alinhamentos de sequência de proteínas e sequências de DNA de promotores alvo derivados de um banco de dados de genes regulados positivamente por TALEs . O segundo grupo (Boch) deduziu o código por meio de análise molecular do efetor TAL AvrBs3 e sua sequência de DNA alvo no promotor de um gene de pimenta ativado por AvrBs3. O código validado experimentalmente entre a sequência RVD e a base de DNA alvo pode ser expresso da seguinte forma:
| Resíduo | Base | Notas | Referências |
|---|---|---|---|
| NI | UMA | ||
| HD | C | Não é 5-metil-C | |
| NG | T, 5 m C | ||
| NN | R | Purina: G ou A | |
| NS | N | Algum | |
| NK | G | Atividade TALEN reduzida se usado exclusivamente | |
| NH | G |
Genes alvo
Os efetores TAL podem induzir genes de suscetibilidade que são membros da família de genes NODULIN3 (N3). Esses genes são essenciais para o desenvolvimento da doença. No arroz, dois genes, Os-8N3 e Os-11N3, são induzidos por efetores de TAL. Os-8N3 é induzido por PthXo1 e Os-11N3 é induzido por PthXo3 e AvrXa7. Existem duas hipóteses sobre as funções possíveis para as proteínas N3:
- Eles estão envolvidos no transporte de cobre, resultando na desintoxicação do ambiente para as bactérias. A redução do nível de cobre facilita o crescimento bacteriano.
- Eles estão envolvidos no transporte de glicose, facilitando o fluxo de glicose. Este mecanismo fornece nutrientes para as bactérias e estimula o crescimento e a virulência do patógeno
Efetores de engenharia TAL
Esta correspondência simples entre aminoácidos em efetores de TAL e bases de DNA em seus locais de destino os torna úteis para aplicações de engenharia de proteínas. Numerosos grupos projetaram efetores TAL artificiais, capazes de reconhecer novas sequências de DNA em uma variedade de sistemas experimentais. Esses efetores TAL projetados foram usados para criar fatores de transcrição artificiais que podem ser usados para direcionar e ativar ou reprimir genes endógenos em tomate , Arabidopsis thaliana e células humanas.
Construções genéticas para codificar proteínas baseadas em efetoras TAL podem ser feitas usando síntese de gene convencional ou montagem modular. Um kit de plasmídeo para montar TALEN personalizado e outras construções efetoras de TAL está disponível no repositório público sem fins lucrativos Addgene . As páginas da Web que fornecem acesso a software público, protocolos e outros recursos para aplicativos de direcionamento de DNA efetor TAL incluem o TAL Effector-Nucleotide Targeter e taleffectors.com .
Formulários
Os efetores TAL projetados também podem ser fundidos ao domínio de clivagem de FokI para criar nucleases efetoras de TAL (TALEN) ou a meganucleases (nucleases com locais de reconhecimento mais longos) para criar "megaTALs". Essas fusões compartilham algumas propriedades com as nucleases de dedo de zinco e podem ser úteis para aplicações de engenharia genética e terapia gênica .
Abordagens baseadas em TALEN são usadas nos campos emergentes de edição de genes e engenharia de genoma . As fusões de TALEN mostram atividade em um ensaio baseado em levedura, em genes de levedura endógenos, em um ensaio repórter de planta, em um gene de planta endógeno, em genes de peixe-zebra endógenos , em um gene de rato endógeno e em genes humanos endógenos. O gene HPRT1 humano foi direcionado a níveis detectáveis, mas não quantificados. Além disso, os construtos TALEN contendo o domínio de clivagem FokI fundido a uma porção menor do efetor TAL ainda contendo o domínio de ligação ao DNA foram usados para direcionar os genes endógenos NTF3 e CCR5 em células humanas com eficiências de até 25%. As nucleases efetoras TAL também foram usadas para projetar células-tronco embrionárias humanas e células-tronco pluripotentes induzidas (IPSCs) e para eliminar o gene ben-1 endógeno em C. elegans .
A modificação da junção da extremidade não homóloga induzida por TALE foi usada para produzir nova resistência a doenças em arroz.
Veja também
- Proteína de ligação ao DNA
- Domínio de ligação de DNA
- Motivo de sequência
- Curtidas no TALE
- Dedo de zinco
- Nuclease de dedo de zinco
Referências
Leitura adicional
- Schornack S, Boch J (2010). “Desvendando um enigma de 20 anos” . IS-MPMI Reporter . International Society for Molecular Plant-Microbe Interactions (1): 3-4.
- Goodsell D (dezembro de 2014). "Efetores TAL" . Molécula do mês do Protein Data Bank (PDB) . Colaborador de Pesquisa em Bioinformática Estrutural (RCSB). doi : 10.2210 / rcsb_pdb / mom_2014_12 .
links externos
- "Um recurso abrangente e publicamente disponível para a tecnologia projetada de efetores TAL" . TALengineering.org .
-
"Grupo de notícias TALengineering" .
Grupo de notícias para discussão da tecnologia projetada de efetores TAL
- "Um recurso aberto para construções efetoras TAL" . www.taleffectors.com .
