Alanine racemase - Alanine racemase
| alanina racemase | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 Homotetrâmero de alanina racemase, Oenococcus oeni
| |||||||||
| Identificadores | |||||||||
| EC nº | 5.1.1.1 | ||||||||
| CAS no. | 9024-06-0 | ||||||||
| Bancos de dados | |||||||||
| IntEnz | Vista IntEnz | ||||||||
| BRENDA | Entrada BRENDA | ||||||||
| ExPASy | NiceZyme view | ||||||||
| KEGG | Entrada KEGG | ||||||||
| MetaCyc | via metabólica | ||||||||
| PRIAM | perfil | ||||||||
| Estruturas PDB | RCSB PDB PDBe PDBsum | ||||||||
| Ontologia Genética | AmiGO / QuickGO | ||||||||
| |||||||||
| Ala_racemase_N | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 o 1,45Å uma estrutura de cristal de alanina racemase de uma bactéria patogênica, pseudomonas aeruginosa, contém as formas de aldimina interna e externa
| |||||||||
| Identificadores | |||||||||
| Símbolo | Ala_racemase_N | ||||||||
| Pfam | PF01168 | ||||||||
| Clã Pfam | CL0036 | ||||||||
| InterPro | IPR001608 | ||||||||
| PRÓSITO | PDOC00332 | ||||||||
| SCOP2 | 1sft / SCOPe / SUPFAM | ||||||||
| |||||||||
| Ala_racemase_C | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Identificadores | |||||||||
| Símbolo | Ala_racemase_C | ||||||||
| Pfam | PF00842 | ||||||||
| InterPro | IPR011079 | ||||||||
| PRÓSITO | PDOC00332 | ||||||||
| SCOP2 | 1sft / SCOPe / SUPFAM | ||||||||
| |||||||||
Em enzimologia , uma racemase de alanina ( EC 5.1.1.1 ) é uma enzima que catalisa a reação química
- L-alanina D-alanina
Portanto, esta enzima tem um substrato , L-alanina , e um produto , D-alanina .
Esta enzima pertence à família das isomerases , especificamente as racemases e epimerases que atuam em aminoácidos e derivados. O nome sistemático desta classe de enzimas é alanina racemase . Esta enzima também é chamada de L-alanina racemase . Esta enzima participa do metabolismo da alanina e do aspartato e do metabolismo da D- alanina . Ele emprega um cofator , o fosfato de piridoxal . Sabe-se que pelo menos dois compostos, 3-Fluoro-D-alanina e D-Cicloserina inibem esta enzima .
A D-alanina produzida pela alanina racemase é usada para a biossíntese de peptidoglicanos. O peptidoglicano é encontrado nas paredes celulares de todas as bactérias, incluindo muitas que são prejudiciais aos seres humanos. A enzima está ausente em eucariotos superiores, mas é encontrada em todos os procariontes, tornando a alanina racemase um grande alvo para o desenvolvimento de drogas antimicrobianas. A racemase alanina pode ser encontrada em alguns invertebrados.
As bactérias podem ter um (gene alr) ou dois genes alanina racemase. As espécies bacterianas com dois genes para alanina racemase têm um que é continuamente expresso e outro que é induzível, o que torna difícil direcionar os dois genes para estudos de drogas. No entanto, estudos de eliminação mostraram que, sem o gene alr sendo expresso, a bactéria precisaria de uma fonte externa de D-alanina para sobreviver. Portanto, o gene alr é um alvo viável para drogas antimicrobianas.
Estudos estruturais
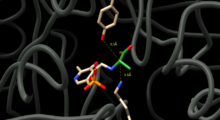
Para catalisar a interconversão de D e L alanina, Alanina racemase deve posicionar resíduos capazes de trocar prótons em ambos os lados do carbono alfa da alanina. Estudos estruturais de complexos enzimáticos-inibidores sugerem que a Tirosina 265 e a Lisina 39 são esses resíduos. O próton alfa do enantiômero L é orientado para Tyr265 e o próton alfa do enantiômero D é orientado para Lys39 (Figura 1).
A distância entre os resíduos de enzima e os enantiômeros é 3,5A e 3,6A, respectivamente. Estudos estruturais de complexos enzimáticos com um análogo sintético de L-alanina, um inibidor de ligação forte e propionato validam ainda que Tyr265 e Lys39 são bases catalíticas para a reação.
Os complexos PLP-L-Ala e PLP-D-Ala são quase sobreponíveis. As regiões que não se sobrepõem são os braços conectados ao anel piridina do PLP e o carbono alfa da alanina. Uma interação entre os átomos de oxigênio fosfato e nitrogênio da piridina com a região 5'fosfopiridoxila de PLP-Ala provavelmente cria uma forte ligação à enzima.
A estrutura da alanina racemase de Bacillus stearothermophilus (Geobacillus stearothermophilus) foi determinada por cristalografia de raios-X com uma resolução de 1,9 A. O monômero alanina racemase é composto por dois domínios, um barril alfa / beta de oito fitas no terminal N, e um domínio C-terminal essencialmente composto de fita beta. Um modelo da estrutura de dois domínios é mostrado na Figura 2. O domínio N-terminal também é encontrado na família de proteínas PROSC (prolina sintetase co-transcrita homóloga bacteriana ), que não são conhecidos por terem atividade de alanina racemase. O cofator de piridoxal 5'-fosfato (PLP) encontra-se dentro e acima da boca do barril alfa / beta e está covalentemente ligado por meio de uma ligação aldimina a um resíduo de lisina , que está no terminal C da primeira fita beta do alfa / barril beta.
Mecanismo proposto
Os mecanismos de reação são difíceis de provar completamente por meio de experimentos. O mecanismo tradicional atribuído a uma reação de racemase de alanina é o de um mecanismo de duas bases com um intermediário de carbanião estabilizado com PLP. PLP é usado como um sumidouro de elétrons para estabilizar a carga negativa resultante da desprotonação do carbono alfa. O mecanismo de base dupla favorece a especificidade da reação em comparação com o mecanismo de base única. O segundo resíduo catalítico é pré-posicionado para doar um próton rapidamente após a formação de um intermediário carbaniônico, reduzindo assim a chance de ocorrerem reações alternativas. Existem dois conflitos potenciais com este mecanismo tradicional, conforme identificado por Watanabe et al. Primeiro, Arg219 forma uma ligação de hidrogênio com nitrogênio de piridina de PLP. O grupo arginina tem um pKa de cerca de 12,6 e, portanto, é improvável que protone a piridina. Normalmente, em reações de PLP, um resíduo de aminoácido ácido, como um grupo de ácido carboxílico, com um pKa de cerca de 5, protona o anel de piridina. A protonação do nitrogênio da piridina permite que o nitrogênio aceite carga negativa adicional. Portanto, devido ao Arg219, o intermediário carbânion estabilizado com PLP é menos provável de se formar. Outro problema identificado foi a necessidade de outro resíduo básico para retornar Lys39 e Tyr265 de volta às suas formas protonadas e não protonadas para L-alanina e vice-versa para D-alanina. Watanabe et al. não encontraram resíduos de aminoácidos ou moléculas de água, além do grupo carboxilato de PLP-Ala, próximos o suficiente (dentro de 4.5A) para protonar ou desprotonar Lys ou Tyr. Isso é mostrado na Figura 3.
Com base nas estruturas cristalinas de N- (5'-fosfopiridoxil) L-alanina (PKP-L-Ala (e N- (5'-fosfopiridoxil) D-alanina (PLP-D-Ala)
Watanabe et al. propôs um mecanismo alternativo em 2002, como visto na figura 4. Neste mecanismo os átomos de oxigênio carboxilato de PLP-Ala participam diretamente da catálise mediando a transferência de prótons entre Lys39 e Tyr265. A estrutura de cristalização identificou que o oxigênio carboxilato de PLP-L-Ala para o OH de Tyr265 era apenas 3,6A e o oxigênio carboxilato de PLP-L-Ala para o nitrogênio de Lys39 era apenas 3,5A. Portanto, ambos estavam próximos o suficiente para causar uma reação.
Este mecanismo é suportado por mutações de Arg219. Mutações alterando Arg219 para um resultado de carboxilato na detecção de um intermediário quinonoide, enquanto nenhum foi detectado com arginina. O intermediário arginina tem muito mais energia livre, é mais instável, do que os mutantes de resíduos ácidos. A desestabilização do intermediário promove a especificidade da reação.
Referências
Leitura adicional
- MARR AG, WILSON PW (1954). "A racemase de alanina de Brucella abortus". Arco. Biochem. Biophys . 49 (2): 424–33. doi : 10.1016 / 0003-9861 (54) 90211-8 . PMID 13159289 .
- Wood WA (1955). "Racemases de aminoácidos". Methods Enzymol . Métodos em Enzimologia. 2 : 212–217. doi : 10.1016 / S0076-6879 (55) 02189-7 . ISBN 9780121818029.
- WOOD WA, GUNSALUS IC (1951). "Formação de D-Alanina; uma racemase em Streptococcus faecalis". J. Biol. Chem . 190 (1): 403–16. PMID 14841188 .