Sentido (biologia molecular) - Sense (molecular biology)
Em biologia molecular e genética , o sentido de uma molécula de ácido nucleico , particularmente de uma fita de DNA ou RNA , refere-se à natureza das funções da fita e seu complemento na especificação de uma sequência de aminoácidos . Dependendo do contexto, o sentido pode ter significados ligeiramente diferentes. Por exemplo, a fita com sentido negativo de DNA é equivalente à fita modelo, enquanto a fita com sentido positivo é a fita não modelo cuja sequência de nucleotídeos é equivalente à sequência do transcrito do mRNA.
Sentido de DNA
Devido à natureza complementar do emparelhamento de bases entre os polímeros de ácido nucléico, uma molécula de DNA de fita dupla será composta de duas fitas com sequências que são complementos reversos uma da outra. Para ajudar os biólogos moleculares a identificar especificamente cada fita individualmente, as duas fitas são geralmente diferenciadas como a fita "sentido" e a fita "anti-sentido". Uma fita individual de DNA é referida como sentido positivo (também positivo (+) ou simplesmente sentido ) se sua sequência de nucleotídeos corresponder diretamente à sequência de um transcrito de RNA que é traduzido ou traduzível em uma sequência de aminoácidos (desde que qualquer bases de timina na sequência de DNA são substituídas por bases de uracila na sequência de RNA). A outra fita da molécula de DNA de fita dupla é chamada de sentido negativo (também negativo (-) ou antissentido ) e é complementarmente reversa à fita de sentido positivo e ao transcrito de RNA. Na verdade, é a fita antisense que é usada como o modelo a partir do qual RNA polimerases constroem o transcrito de RNA, mas o emparelhamento de base complementar pelo qual ocorre a polimerização de ácido nucleico significa que a sequência do transcrito de RNA parecerá idêntica à fita sense, à parte do uso do transcrito de RNA de uracila em vez de timina.
Às vezes, as frases fita de codificação e fita modelo são encontradas no lugar de sentido e anti-sentido, respectivamente, e no contexto de uma molécula de DNA de fita dupla, o uso desses termos é essencialmente equivalente. No entanto, a fita de codificação / detecção nem sempre precisa conter um código que é usado para fazer uma proteína; ambos os RNAs codificadores de proteínas e não codificantes podem ser transcritos.
Os termos "sentido" e "anti-sentido" são relativos apenas ao transcrito de RNA específico em questão, e não à fita de DNA como um todo. Em outras palavras, qualquer fita de DNA pode servir como fita sense ou antisense. A maioria dos organismos com genomas suficientemente grandes faz uso de ambas as fitas, com cada fita funcionando como a fita modelo para diferentes transcritos de RNA em diferentes lugares ao longo da mesma molécula de DNA. Em alguns casos, os transcritos de RNA podem ser transcritos em ambas as direções (ou seja, em qualquer fita) a partir de uma região promotora comum , ou ser transcritos de dentro dos íntrons em qualquer fita (ver "ambisense" abaixo).
DNA anti-sentido
A fita com sentido de DNA se parece com o transcrito do RNA mensageiro (mRNA) e pode, portanto, ser usada para ler a sequência de códons esperada que será usada durante a tradução (síntese de proteína) para construir uma sequência de aminoácidos e, em seguida, uma proteína. Por exemplo, a sequência "ATG" dentro de uma fita com sentido de DNA corresponde a um códon "AUG" no mRNA, que codifica para o aminoácido metionina . No entanto, a própria cadeia de DNA sense não é usada como molde para o mRNA; é a fita antisense de DNA que serve como fonte para o código da proteína, pois, com bases complementares à fita sense de DNA, é usada como molde para o mRNA. Uma vez que a transcrição resulta em um produto de RNA complementar à fita modelo de DNA, o mRNA é complementar à fita antisense de DNA.
Assim, um tripleto de base 3′-TAC-5 ′ na fita antisense de DNA (complementar ao 5′-ATG-3 ′ da fita de DNA sense) é usado como o modelo que resulta em um 5′-AUG-3 ′ tripleto de base no mRNA. A fita sense de DNA terá o tripleto ATG, que se parece com o tripleto AUG do mRNA, mas não será usado para fazer metionina porque não será usado diretamente para fazer mRNA. A fita de DNA sense é chamada de fita "sense" não porque será usada para fazer proteínas (não será), mas porque tem uma sequência que corresponde diretamente à sequência do códon do RNA. Por essa lógica, a própria transcrição de RNA é algumas vezes descrita como "sentido".
Exemplo com DNA de fita dupla
- Fita de DNA 1: fita anti-sentido (transcrita para) → fita de RNA (sentido)
- Fita de DNA 2: fita de sentido
Algumas regiões dentro de um código de molécula de ADN de cadeia dupla para genes , que são normalmente as instruções especificando a ordem em que os aminoácidos são montados para fazer proteínas, bem como sequências reguladoras, splicing locais, não-codificante intrões , e outros produtos do gene . Para que uma célula use essa informação, uma fita de DNA serve como molde para a síntese de uma fita complementar de RNA . A fita de DNA transcrita é chamada de fita modelo, com sequência antisense, e o transcrito de mRNA produzido a partir dela é chamado de sequência sense (o complemento do antisense). A fita de DNA não transcrita, complementar à fita transcrita, também é considerada como tendo sequência de sentido; tem a mesma sequência de sentido que o transcrito do mRNA (embora as bases T no DNA sejam substituídas por bases U no RNA).
| 3′CGCTATAGCGTTT 5 ′ | Fita antisense de DNA (molde / não codificante) | Usado como modelo para transcrição. |
| 5′GCGATATCGCAAA 3 ′ | Cadeia com sentido de DNA (não modelo / codificação) | Complementar à vertente do molde. |
| 5′GCGAUAUCGCAAA 3 ′ | transcrição sentido do mRNA | Fita de RNA que é transcrita da fita não codificadora (molde / antisense). Nota 1 : Exceto pelo fato de que todas as timas agora são uracilos ( T → U ), é complementar à fita de DNA não codificante (modelo / antisense) e idêntica à fita de DNA codificante (não modelo / sentido) . |
| 3′CGCUAUAGCGUUU 5 ′ | transcrito antisense de mRNA | Cadeia de RNA que é transcrita da cadeia de codificação (não-modelo / sentido). Nota: Exceto pelo fato de que todas as timas agora são uracilos ( T → U ), é complementar à fita de DNA codificante (não-modelo / sentido) e idêntica à fita de DNA não-codificante (molde / anti-sentido). |
Os nomes atribuídos a cada fita dependem, na verdade, da direção em que você está escrevendo a sequência que contém as informações das proteínas (as informações de "sentido"), e não de qual fita é descrita como "no topo" ou "na parte inferior" (que é arbitrário). A única informação biológica que é importante para as fitas de marcação são as localizações relativas do grupo terminal 5 'fosfato e o grupo terminal 3' hidroxila (nas extremidades da fita ou sequência em questão), porque essas extremidades determinam a direção da transcrição e tradução. Uma sequência escrita 5′-CGCTAT-3 ′ é equivalente a uma sequência escrita 3′-TATCGC-5 ′, desde que as extremidades 5 'e 3' sejam anotadas. Se as extremidades não são rotuladas, a convenção é assumir que ambas as sequências são escritas na direção 5′ para 3 ′. A "fita Watson" refere-se à fita superior de 5′ para 3 ′ (5 ′ → 3 ′), enquanto a "fita de Crick" se refere à fita inferior de 5′ para 3 ′ (3 ′ ← 5 ′). Ambas as fitas Watson e Crick podem ser fitas sense ou antisense, dependendo do produto do gene específico feito a partir delas.
Por exemplo, a notação "YEL021W", um alias do gene URA3 usado no banco de dados do National Center for Biotechnology Information (NCBI), denota que esse gene está no 21º quadro de leitura aberta (ORF) do centrômero do braço esquerdo ( L) do cromossomo de levedura (Y) número V (E), e que a fita codificadora da expressão é a fita Watson (W). "YKL074C" denota a 74ª ORF à esquerda do centrômero do cromossomo XI e que a fita de codificação é a fita de Crick (C). Outro termo confuso que se refere às fitas "Mais" e "Menos" também é amplamente utilizado. Quer a fita seja com sentido (positiva) ou anti-sentido (negativa), a sequência de consulta padrão no alinhamento do NCBI BLAST é a fita "Mais".
Ambisense
Um genoma de fita simples que é usado tanto nas capacidades de sentido positivo quanto negativo é considerado ambissenso . Alguns vírus têm genomas ambisense. Os Bunyavírus têm três fragmentos de RNA de fita simples (ssRNA), alguns deles contendo seções de sentido positivo e negativo; arenavírus também são vírus ssRNA com um genoma ambisense, pois eles têm três fragmentos que são principalmente de sentido negativo, exceto por parte das extremidades 5 'dos segmentos grandes e pequenos de seu genoma.
RNA anti-sentido
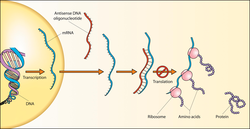
Uma sequência de RNA que é complementar a um transcrito de mRNA endógeno é algumas vezes chamada de " RNA antisense ". Em outras palavras, é uma fita não codificadora complementar à sequência codificadora do RNA; isso é semelhante ao RNA viral de sentido negativo. Quando o mRNA forma um duplex com uma sequência complementar de RNA antisense, a tradução é bloqueada. Este processo está relacionado à interferência de RNA . As células podem produzir moléculas de RNA antisense naturalmente, chamadas microRNAs , que interagem com moléculas de mRNA complementares e inibem sua expressão . O conceito também foi explorado como uma técnica de biologia molecular, por meio da introdução artificial de um transgene que codifica o RNA antisense para bloquear a expressão de um gene de interesse. O ARN anti-sentido marcado radioactivamente ou fluorescentemente pode ser utilizado para mostrar o nível de transcrição de genes em vários tipos de células.
Alguns tipos estruturais antisense alternativos foram aplicados experimentalmente como terapia antisense . Nos Estados Unidos, a Food and Drug Administration (FDA) aprovou os oligonucleotídeos antisense fosforotioato fomivirsen (Vitravene) e mipomersen (Kynamro) para uso terapêutico em humanos.
Sentido de RNA em vírus
Em virologia , o termo "sentido" tem um significado ligeiramente diferente. O genoma de um vírus de RNA pode ser considerado de sentido positivo , também conhecido como "fita positiva", ou sentido negativo , também conhecido como "fita negativa". Na maioria dos casos, os termos "sentido" e "fita" são usados indistintamente, tornando termos como "fita positiva" equivalente a "sentido positivo" e "fita mais" equivalente a "sentido mais". O fato de um genoma viral ser de sentido positivo ou negativo pode ser usado como base para a classificação de vírus.
Senso positivo
O RNA viral de sentido positivo ( 5 ′ -para- 3 ′ ) significa que uma sequência específica de RNA viral pode ser traduzida diretamente em proteínas virais (por exemplo, aquelas necessárias para a replicação viral). Portanto, em vírus de RNA de sentido positivo, o genoma de RNA viral pode ser considerado mRNA viral e pode ser imediatamente traduzido pela célula hospedeira. Ao contrário do RNA de sentido negativo, o RNA de sentido positivo tem o mesmo sentido que o mRNA. Alguns vírus (por exemplo, Coronaviridae ) têm genomas de sentido positivo que podem atuar como mRNA e ser usados diretamente para sintetizar proteínas sem a ajuda de um intermediário de RNA complementar. Por causa disso, esses vírus não precisam ter uma RNA polimerase empacotada no vírion - a RNA polimerase será uma das primeiras proteínas produzidas pela célula hospedeira, uma vez que é necessária para que o genoma do vírus seja replicado.
Sentido negativo
O RNA viral de sentido negativo (3′-a-5 ′) é complementar ao mRNA viral, portanto, um RNA de sentido positivo deve ser produzido por uma RNA polimerase dependente de RNA antes da tradução. Como o DNA, o RNA de sentido negativo tem uma sequência de nucleotídeos complementar ao mRNA que ele codifica; também como o DNA, esse RNA não pode ser traduzido em proteína diretamente. Em vez disso, deve primeiro ser transcrito em um RNA de sentido positivo que atua como um mRNA. Alguns vírus (por exemplo, vírus influenza ) têm genomas de sentido negativo e, portanto, devem carregar uma RNA polimerase dentro do vírion.
Oligonucleotídeos antisense
O silenciamento de genes pode ser conseguido introduzindo nas células um curto "oligonucleotídeo antisense" que é complementar a um alvo de RNA. Este experimento foi feito pela primeira vez por Zamecnik e Stephenson em 1978 e continua a ser uma abordagem útil, tanto para experimentos de laboratório como potencialmente para aplicações clínicas ( terapia antisense ). Vários vírus, como vírus influenza, vírus sincicial respiratório (RSV) e coronavírus SARS (SARS-CoV), foram direcionados usando oligonucleotídeos antisense para inibir sua replicação em células hospedeiras.
Se o oligonucleotídeo antisense contiver um trecho de DNA ou um mimetizador de DNA (DNA fosforotioato, 2'F-ANA ou outros), ele pode recrutar RNase H para degradar o RNA alvo. Isso torna o mecanismo de silenciamento de genes catalítico. O RNA de fita dupla também pode atuar como um agente antisense catalítico dependente de enzima por meio da via de RNAi / siRNA , envolvendo o reconhecimento de mRNA alvo por meio de pareamento de fita sentido-antisense seguido pela degradação de mRNA alvo pelo complexo de silenciamento induzido por RNA (RISC). O sistema hok / sok do plasmídeo R1 fornece ainda outro exemplo de um processo de regulação antisense dependente de enzima por meio da degradação enzimática do duplex de RNA resultante.
Outros mecanismos antisense não são dependentes de enzimas, mas envolvem o bloqueio estérico de seu RNA alvo (por exemplo, para evitar a tradução ou para induzir splicing alternativo). Os mecanismos antisense de bloqueio estérico geralmente usam oligonucleotídeos fortemente modificados. Uma vez que não há necessidade de reconhecimento de RNase H, isso pode incluir produtos químicos como 2'-O-alquil, ácido nucleico de peptídeo (PNA), ácido nucleico bloqueado (LNA) e oligômeros de Morfolino .
Veja também
- Terapia anti-sentido
- Direcionalidade (biologia molecular)
- Tabela de códons de DNA
- Vírus de RNA
- Transcrição (genética)
- Tradução (genética)
- Replicação viral