microRNA - microRNA
Um microRNA ( miRNA abreviado ) é uma pequena molécula de RNA não codificante de fita simples (contendo cerca de 22 nucleotídeos ) encontrada em plantas, animais e alguns vírus, que funciona no silenciamento do RNA e na regulação pós-transcricional da expressão gênica . Os miRNAs funcionam via emparelhamento de bases com sequências complementares dentro das moléculas de mRNA . Como resultado, essas moléculas de mRNA são silenciadas , por um ou mais dos seguintes processos: (1) clivagem da fita de mRNA em duas partes, (2) desestabilização do mRNA através do encurtamento de sua cauda poli (A) , e ( 3) tradução menos eficiente do mRNA em proteínas pelos ribossomos .
Os miRNAs se assemelham aos pequenos RNAs interferentes (siRNAs) da via de interferência do RNA (RNAi) , exceto que os miRNAs derivam de regiões de transcritos de RNA que se dobram para formar grampos curtos, enquanto os siRNAs derivam de regiões mais longas de RNA de fita dupla . O genoma humano pode codificar mais de 1900 miRNAs, embora análises mais recentes sugiram que o número está mais próximo de 2.300. No entanto, apenas cerca de 500 microRNAs humanos representam miRNA genuíno no banco de dados de genes de miRNA com curadoria manual MirGeneDB .
Os miRNAs são abundantes em muitos tipos de células de mamíferos e como miRNAs circulantes extracelulares . Os miRNAs circulantes são liberados nos fluidos corporais, incluindo sangue e líquido cefalorraquidiano, e têm o potencial de estar disponíveis como biomarcadores em uma série de doenças. Os miRNAs parecem ter como alvo cerca de 60% dos genes de humanos e outros mamíferos. Muitos miRNAs são conservados evolutivamente, o que implica que eles têm funções biológicas importantes. Por exemplo, 90 famílias de miRNAs foram conservadas desde pelo menos o ancestral comum de mamíferos e peixes, e a maioria desses miRNAs conservados têm funções importantes, conforme mostrado por estudos em que genes para um ou mais membros de uma família foram eliminados Em ratos.
História
O primeiro miRNA foi descoberto no início de 1990. No entanto, os miRNAs não foram reconhecidos como uma classe distinta de reguladores biológicos até o início dos anos 2000. A pesquisa de miRNAs revelou diferentes conjuntos de miRNAs expressos em diferentes tipos de células e tecidos e vários papéis para miRNAs no desenvolvimento de plantas e animais e em muitos outros processos biológicos. Expressão de miRNA aberrante está implicada em estados de doença. As terapias baseadas em MiRNA estão sob investigação.
O primeiro miRNA foi descoberto em 1993 por um grupo liderado por Ambros e incluindo Lee e Feinbaum. No entanto, uma visão adicional sobre seu modo de ação exigiu um trabalho publicado simultaneamente pela equipe de Ruvkun , incluindo Wightman e Ha. Esses grupos publicaram artigos consecutivos sobre o gene lin-4 , que era conhecido por controlar o tempo de desenvolvimento larval de C. elegans ao reprimir o gene lin-14 . Quando Lee et al. isolaram o miRNA lin-4 , eles descobriram que em vez de produzir um mRNA que codifica uma proteína, ele produziu RNAs não codificantes curtos , um dos quais era um RNA de ~ 22 nucleotídeos que continha sequências parcialmente complementares a múltiplas sequências na UTR 3 ' do mRNA de lin-14 . Esta complementaridade foi proposta para inibir a tradução do mRNA de lin-14 na proteína LIN-14. Na época, pensava -se que o pequeno RNA lin-4 era uma idiossincrasia do nematóide .
Em 2000, um segundo pequeno RNA foi caracterizado: let-7 RNA, que reprime lin-41 para promover uma transição de desenvolvimento posterior em C. elegans . O RNA let-7 foi encontrado para ser conservado em muitas espécies, levando à sugestão de que o RNA let-7 e "pequenos RNAs temporais" adicionais podem regular o tempo de desenvolvimento em diversos animais, incluindo humanos.
Um ano depois, descobriu -se que os RNAs lin-4 e let-7 fazem parte de uma grande classe de pequenos RNAs presentes em C. elegans , Drosophila e células humanas. Os muitos RNAs desta classe se assemelhavam aos RNAs lin-4 e let-7 , exceto que seus padrões de expressão eram geralmente inconsistentes com um papel na regulação do tempo de desenvolvimento. Isso sugeriu que a maioria pode funcionar em outros tipos de vias regulatórias. Nesse ponto, os pesquisadores começaram a usar o termo "microRNA" para se referir a essa classe de pequenos RNAs reguladores.
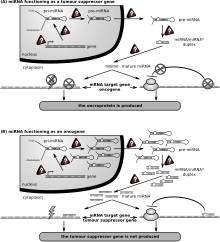
A primeira doença humana associada à desregulação de miRNAs foi a leucemia linfocítica crônica . Nesse distúrbio, os miRNAs têm um papel duplo, atuando como supressores de tumor e oncogenes.
Nomenclatura
Sob um sistema de nomenclatura padrão, os nomes são atribuídos a miRNAs confirmados experimentalmente antes da publicação. O prefixo "miR" é seguido por um hífen e um número, o último geralmente indicando a ordem de nomenclatura. Por exemplo, miR-124 foi nomeado e provavelmente descoberto antes de miR-456. Um "miR-" maiúsculo refere-se à forma madura do miRNA, enquanto o "mir-" não capitalizado se refere ao pré-miRNA e ao pri-miRNA. Os genes que codificam miRNAs também são nomeados usando o mesmo prefixo de três letras de acordo com as convenções da nomenclatura de genes do organismo. Por exemplo, os nomes de genes de miRNAs oficiais em alguns organismos são " mir-1 em C. elegans e Drosophila, Mir-1 em Rattus norvegicus e MIR-25 em humanos.
miRNAs com sequências quase idênticas, exceto por um ou dois nucleotídeos, são anotados com uma letra minúscula adicional. Por exemplo, miR-124a está intimamente relacionado com miR-124b. Por exemplo:
- hsa-miR-181a :aacauucaACgcugucggugAgu
- hsa-miR-181b :aacauucaUUgcugucggugGgu
Pré-miRNAs, pri-miRNAs e genes que levam a miRNAs maduros 100% idênticos, mas que estão localizados em locais diferentes no genoma, são indicados com um sufixo de número de traço adicional. Por exemplo, os pré-miRNAs hsa-mir-194-1 e hsa-mir-194-2 levam a um miRNA maduro idêntico (hsa-miR-194), mas são de genes localizados em diferentes regiões do genoma.
A espécie de origem é designada com um prefixo de três letras, por exemplo, hsa-miR-124 é um miRNA humano ( Homo sapiens ) e oar-miR-124 é um miRNA de ovelha ( Ovis aries ). Outros prefixos comuns incluem "v" para viral (miRNA codificado por um genoma viral) e "d" para miRNA de Drosophila (uma mosca da fruta comumente estudada em pesquisas genéticas).
Quando dois microRNAs maduros se originam de braços opostos do mesmo pré-miRNA e são encontrados em quantidades aproximadamente semelhantes, eles são denotados com um sufixo -3p ou -5p. (No passado, essa distinção também era feita com "s" ( sentido ) e "como" (anti-sentido)). No entanto, o microRNA maduro encontrado em um braço do grampo de cabelo é geralmente muito mais abundante do que o encontrado no outro braço, nesse caso, um asterisco após o nome indica a espécie madura encontrada em níveis baixos do braço oposto de um grampo. Por exemplo, miR-124 e miR-124 * compartilham um hairpin pré-miRNA, mas muito mais miR-124 é encontrado na célula.
Alvos
Os miRNAs de plantas geralmente têm emparelhamento quase perfeito com seus alvos de mRNA, o que induz a repressão gênica por meio da clivagem dos transcritos alvo. Em contraste, miRNAs de animais são capazes de reconhecer seus mRNAs alvo usando apenas 6–8 nucleotídeos (a região da semente) na extremidade 5 'do miRNA, o que não é emparelhamento suficiente para induzir a clivagem dos mRNAs alvo. A regulação combinatória é uma característica da regulação de miRNA em animais. Um determinado miRNA pode ter centenas de diferentes alvos de mRNA, e um determinado alvo pode ser regulado por vários miRNAs.
As estimativas do número médio de RNAs mensageiros únicos que são alvos para repressão por um miRNA típico variam, dependendo do método de estimativa, mas várias abordagens mostram que miRNAs de mamíferos podem ter muitos alvos únicos. Por exemplo, uma análise dos miRNAs altamente conservados em vertebrados mostra que cada um tem, em média, cerca de 400 alvos conservados. Da mesma forma, experimentos mostram que uma única espécie de miRNA pode reduzir a estabilidade de centenas de RNAs mensageiros únicos. Outros experimentos mostram que uma única espécie de miRNA pode reprimir a produção de centenas de proteínas, mas que essa repressão costuma ser relativamente leve (muito menos do que 2 vezes). A primeira doença humana descoberta associada à desregulação de miRNAs foi a leucemia linfocítica crônica . Seguiram-se outras doenças malignas de células B.
Biogênese
Até 40% dos genes de miRNA podem estar nos íntrons ou mesmo nos exons de outros genes. Estes são geralmente, embora não exclusivamente, encontrados em uma orientação sensorial e, portanto, geralmente são regulados junto com seus genes hospedeiros.
O modelo de DNA não é a palavra final sobre a produção de miRNAs maduros: 6% dos miRNAs humanos mostram edição de RNA ( IsomiRs ), a modificação específica do local de sequências de RNA para produzir produtos diferentes daqueles codificados por seu DNA. Isso aumenta a diversidade e o escopo da ação do miRNA além do que está implicado apenas no genoma.
Transcrição
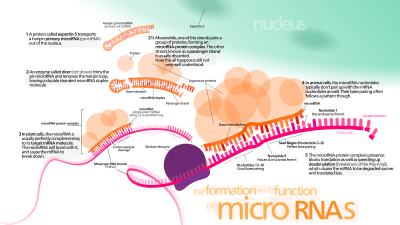
Os genes de miRNA são geralmente transcritos pela RNA polimerase II (Pol II). A polimerase geralmente se liga a um promotor encontrado próximo à sequência de DNA, codificando o que se tornará a alça em gancho do pré-miRNA. O transcrito resultante é coberto com um nucleotídeo especialmente modificado na extremidade 5 ', poliadenilado com múltiplas adenosinas (uma cauda poli (A)) e emendado . Os miRNAs de animais são inicialmente transcritos como parte de um braço de uma haste-alça de RNA de ∼80 nucleotídeos que, por sua vez, faz parte de um precursor de várias centenas de miRNA de comprimento de nucleotídeo denominado um pri-miRNA. Quando um precursor haste-alça é encontrado na UTR 3 ', um transcrito pode servir como um pri-miRNA e um mRNA. A RNA polimerase III (Pol III) transcreve alguns miRNAs, especialmente aqueles com sequências Alu a montante , RNAs de transferência (tRNAs) e unidades promotoras de repetição intercalada em mamíferos (MWIR).
Processamento nuclear

Um único pri-miRNA pode conter de um a seis precursores de miRNA. Essas estruturas de loop em gancho são compostas por cerca de 70 nucleotídeos cada. Cada grampo de cabelo é flanqueado por sequências necessárias para um processamento eficiente.
A estrutura de RNA de fita dupla (dsRNA) dos grampos em um pri-miRNA é reconhecida por uma proteína nuclear conhecida como DiGeorge Syndrome Critical Region 8 (DGCR8 ou "Pasha" em invertebrados), nomeada por sua associação com a Síndrome de DiGeorge . O DGCR8 se associa à enzima Drosha , uma proteína que corta o RNA, para formar o complexo do microprocessador . Neste complexo, DGCR8 orienta o domínio RNase III catalítico de Drosha para liberar grampos de pri-miRNAs por clivagem do RNA de cerca de onze nucleotídeos da base em grampo (um dsRNA helicoidal transforma-se na haste). O produto resultante tem uma saliência de dois nucleotídeos em sua extremidade 3 '; possui grupos 3 'hidroxila e 5' fosfato. Muitas vezes é denominado como um pré-miRNA (precursor-miRNA). Motivos de sequência a jusante do pré-miRNA que são importantes para o processamento eficiente foram identificados.
Os pré-miRNAs que são unidos diretamente dos íntrons, ignorando o complexo do microprocessador, são conhecidos como " Mirtrons ". Originalmente pensado para existir apenas em Drosophila e C. elegans , mirtrons agora foram encontrados em mamíferos.
Até 16% dos pré-miRNAs podem ser alterados por meio da edição de RNA nuclear . Mais comumente, as enzimas conhecidas como adenosina desaminases que atuam no RNA (ADARs) catalisam as transições de adenosina para inosina (A para I). A edição de RNA pode interromper o processamento nuclear (por exemplo, de pri-miR-142, levando à degradação pela ribonuclease Tudor-SN) e alterar processos a jusante, incluindo processamento de miRNA citoplasmático e especificidade de alvo (por exemplo, alterando a região de semente de miR-376 no sistema nervoso central).
Exportação nuclear

Grampos de cabelo pré-miRNA são exportados do núcleo em um processo que envolve o lançador nucleocitoplasmático Exportin-5 . Esta proteína, um membro da família da carioferina , reconhece uma saliência de dois nucleotídeos deixada pela enzima RNase III Drosha na extremidade 3 'do grampo do pré-miRNA. O transporte mediado pela exportina-5 para o citoplasma é dependente de energia, usando trifosfato de guanosina (GTP) ligado à proteína Ran .
Processamento citoplasmático
No citoplasma , o grampo de cabelo pré-miRNA é clivado pela enzima RNase III Dicer . Esta endoribonuclease interage com as extremidades 5 'e 3' do grampo de cabelo e corta a alça que une os braços 3 'e 5', produzindo um miRNA imperfeito: miRNA * duplex com cerca de 22 nucleotídeos de comprimento. O comprimento geral do grampo de cabelo e o tamanho do loop influenciam a eficiência do processamento do Dicer. A natureza imperfeita do par de miRNA: miRNA * também afeta a clivagem. Alguns dos pré-miRNAs ricos em G podem potencialmente adotar a estrutura G-quadruplex como uma alternativa para a estrutura haste-loop canônica. Por exemplo, o pré-miRNA 92b humano adota uma estrutura G-quadruplex que é resistente à clivagem mediada por Dicer no citoplasma . Embora qualquer fita do duplex possa atuar potencialmente como um miRNA funcional, apenas uma fita é geralmente incorporada ao complexo de silenciamento induzido por RNA (RISC), onde o miRNA e seu mRNA alvo interagem.
Embora a maioria dos miRNAs esteja localizada dentro da célula, alguns miRNAs, comumente conhecidos como miRNAs circulantes ou miRNAs extracelulares, também foram encontrados em ambiente extracelular, incluindo vários fluidos biológicos e meios de cultura de células.
Biogênese em plantas
A biogênese de miRNA em plantas difere da biogênese animal principalmente nas etapas de processamento nuclear e exportação. Em vez de ser clivado por duas enzimas diferentes, uma vez dentro e uma vez fora do núcleo, ambas as clivagens do miRNA da planta são realizadas por um homólogo Dicer, denominado Dicer-like1 (DL1). DL1 é expresso apenas no núcleo das células vegetais, o que indica que ambas as reações ocorrem dentro do núcleo. Antes de os duplexes de miRNA: miRNA * da planta serem transportados para fora do núcleo, suas saliências 3 'são metiladas por uma proteína metiltransferase de RNA chamada Hua-Enhancer1 (HEN1). O duplex é então transportado para fora do núcleo para o citoplasma por uma proteína chamada Hasty (HST), um homólogo de Exportin 5, onde se desmontam e o miRNA maduro é incorporado ao RISC.
Complexo de silenciamento induzido por RNA
O miRNA maduro é parte de um complexo de silenciamento induzido por RNA ativo (RISC) contendo Dicer e muitas proteínas associadas. RISC também é conhecido como um complexo de ribonucleoproteína de microRNA (miRNP); Um RISC com miRNA incorporado é algumas vezes referido como "miRISC".
O processamento Dicer do pré-miRNA é pensado para ser acoplado ao desenrolamento do duplex. Geralmente, apenas uma fita é incorporada ao miRISC, selecionada com base em sua instabilidade termodinâmica e pareamento de bases mais fraco na extremidade 5 'em relação à outra fita. A posição da laçada da haste também pode influenciar a escolha do fio. A outra vertente, chamada vertente passageira devido aos seus níveis mais baixos no estado estacionário, é indicada com um asterisco (*) e normalmente está degradada. Em alguns casos, ambas as fitas do duplex são viáveis e se tornam miRNA funcionais que têm como alvo diferentes populações de mRNA.
Membros da família de proteínas Argonaute (Ago) são centrais para a função RISC. Argonautos são necessários para o silenciamento induzido por miRNA e contêm dois domínios de ligação de RNA conservados: um domínio PAZ que pode ligar a extremidade 3 'de fita simples do miRNA maduro e um domínio PIWI que se assemelha estruturalmente à ribonuclease-H e funciona para interagir com o 5' final do fio guia. Eles se ligam ao miRNA maduro e o orientam para interação com um mRNA alvo. Alguns argonautas, por exemplo o Ago2 humano, clivam transcritos alvo diretamente; argonautas também podem recrutar proteínas adicionais para atingir a repressão translacional. O genoma humano codifica oito proteínas argonautas divididas por semelhanças de sequência em duas famílias: AGO (com quatro membros presentes em todas as células de mamíferos e chamados de E1F2C / hAgo em humanos) e PIWI (encontrada na linhagem germinativa e células-tronco hematopoiéticas).
Componentes RISC adicionais incluem TRBP [proteína de ligação de RNA de resposta transativante (TAR) do vírus da imunodeficiência humana (HIV)], PACT (ativador de proteína da proteína quinase induzida por interferon ), o complexo SMN, proteína de retardo mental frágil X (FMRP), Tudor estafilocócica proteína contendo domínio de nuclease (Tudor-SN), a suposta DNA helicase MOV10 e o motivo de reconhecimento de RNA contendo a proteína TNRC6B .
Modo de silenciamento e loops regulatórios
O silenciamento do gene pode ocorrer por meio da degradação do mRNA ou impedindo que o mRNA seja traduzido. Por exemplo, miR16 contém uma sequência complementar ao elemento rico em AU encontrado na 3'UTR de muitos mRNAs instáveis, como TNF alfa ou GM-CSF . Foi demonstrado que dada complementaridade completa entre o miRNA e a sequência de mRNA alvo, o Ago2 pode clivar o mRNA e levar à degradação direta do mRNA. Na ausência de complementaridade, o silenciamento é obtido evitando a tradução. A relação de miRNA e seu mRNA alvo pode ser baseada na regulação negativa simples de um mRNA alvo, mas parece que um cenário comum é o uso de um " loop feed-forward coerente ", "loop de feedback negativo mútuo" (também denominado loop negativo duplo) e "loop de feedback positivo / feed-forward". Alguns miRNAs funcionam como buffers de mudanças de expressão gênica aleatórias decorrentes de eventos estocásticos na transcrição, tradução e estabilidade de proteínas. Tal regulação é tipicamente alcançada em virtude de loops de feedback negativo ou loop feed-forward incoerente que desacopla a saída da proteína da transcrição do mRNA.
Rotatividade
A renovação de miRNA maduro é necessária para mudanças rápidas nos perfis de expressão de miRNA. Durante a maturação do miRNA no citoplasma, acredita-se que a captação pela proteína Argonaute estabilize a fita guia, enquanto a fita oposta (* ou "passageira") é preferencialmente destruída. No que foi chamado de estratégia "Use ou perca", Argonaute pode preferencialmente reter miRNAs com muitos alvos sobre miRNAs com poucos ou nenhum alvo, levando à degradação das moléculas não-alvo.
Decaimento de madura miARNs em Caenorhabditis elegans é mediada pela 3' 5'-para- exoribonuclease XRN2 , também conhecido como Rat1p. Em plantas, os membros da família SDN (small RNA degrading nuclease) degradam miRNAs na direção oposta (3'-a-5 '). Enzimas semelhantes são codificadas em genomas de animais, mas seus papéis não foram descritos.
Várias modificações de miRNA afetam a estabilidade do miRNA. Conforme indicado pelo trabalho no organismo modelo Arabidopsis thaliana (thale cress), miRNAs de plantas maduras parecem ser estabilizados pela adição de metilas na extremidade 3 '. Os grupos metil conjugados 2'-O bloqueiam a adição de resíduos de uracila (U) pelas enzimas uridiltransferase , uma modificação que pode estar associada à degradação de miRNA. No entanto, a uridilação também pode proteger alguns miRNAs; as consequências desta modificação não são completamente compreendidas. A uridilação de alguns miRNAs de animais foi relatada. Ambos os miRNAs de plantas e animais podem ser alterados pela adição de resíduos de adenina (A) à extremidade 3 'do miRNA. Um A extra adicionado ao final do miR-122 de mamífero , um miRNA enriquecido com fígado importante na hepatite C , estabiliza a molécula e os miRNAs da planta que terminam com um resíduo de adenina têm taxas de decomposição mais lentas.
Funções celulares

A função dos miRNAs parece estar na regulação gênica. Para tanto, um miRNA é complementar a uma parte de um ou mais RNAs mensageiros (mRNAs). Os miRNAs de animais são geralmente complementares a um local na UTR 3 ', enquanto os miRNAs de plantas são geralmente complementares às regiões codificantes de mRNAs. O emparelhamento de bases perfeito ou quase perfeito com o RNA alvo promove a clivagem do RNA. Este é o principal modo de miRNAs de plantas. Nos animais, as combinações são imperfeitas.
Para que microRNAs parcialmente complementares reconheçam seus alvos, os nucleotídeos 2-7 do miRNA (sua 'região de semente') devem ser perfeitamente complementares. Os miRNAs de animais inibem a tradução de proteínas do mRNA alvo (isso está presente, mas menos comum em plantas). MicroRNAs parcialmente complementares também podem acelerar a deadenilação , fazendo com que os mRNAs sejam degradados mais cedo. Embora a degradação do mRNA direcionado ao miRNA seja bem documentada, se a repressão translacional é ou não realizada por meio da degradação do mRNA, inibição da tradução ou uma combinação das duas é um debate acalorado. Trabalhos recentes em miR-430 em peixe - zebra, bem como em bantam-miRNA e miR-9 em células cultivadas de Drosophila , mostram que a repressão translacional é causada pela interrupção do início da tradução , independente da deadenilação do mRNA.
Os miRNAs ocasionalmente também causam modificação de histonas e metilação de DNA de sítios promotores , o que afeta a expressão de genes-alvo.
Nove mecanismos de ação do miRNA são descritos e montados em um modelo matemático unificado:
- Inibição de iniciação Cap-40S;
- Inibição de união da unidade ribossomal 60S;
- Inibição de alongamento;
- Queda de ribossomo (término prematuro);
- Degradação de proteína nascente co-translacional;
- Seqüestro em corpos P;
- decaimento do mRNA (desestabilização);
- clivagem de mRNA;
- Inibição da transcrição através da reorganização da cromatina mediada por microRNA seguida de silenciamento do gene.
Freqüentemente, é impossível discernir esses mecanismos usando dados experimentais sobre taxas de reação estacionárias. No entanto, eles são diferenciados na dinâmica e têm diferentes assinaturas cinéticas .
Ao contrário dos microRNAs de plantas, os microRNAs de animais têm como alvo diversos genes. No entanto, os genes envolvidos em funções comuns a todas as células, como a expressão gênica, têm relativamente menos sítios alvo de microRNA e parecem estar sob seleção para evitar o direcionamento por microRNAs.
O dsRNA também pode ativar a expressão gênica , um mecanismo que foi denominado "ativação gênica induzida por RNA pequeno" ou RNAa . Os dsRNAs que têm como alvo os promotores de genes podem induzir ativação transcricional potente de genes associados. Isso foi demonstrado em células humanas usando dsRNAs sintéticos denominados pequenos RNAs ativadores ( saRNAs ), mas também foi demonstrado para microRNAs endógenos.
As interações entre microRNAs e sequências complementares em genes e até mesmo pseudogenes que compartilham homologia de sequência são consideradas um canal traseiro de comunicação regulando os níveis de expressão entre genes parálogos (genes com uma estrutura semelhante indicando divergência de um gene ancestral comum). Dado o nome de "RNAs endógenos competidores" ( ceRNAs ), esses microRNAs se ligam a "elementos de resposta de microRNA" em genes e pseudogenes e podem fornecer outra explicação para a persistência de DNA não codificador.
Algumas pesquisas mostram que a carga de mRNA de exossomos pode ter um papel na implantação, eles podem travar uma adesão entre o trofoblasto e o endométrio ou apoiar a adesão regulando para baixo ou para cima a expressão de genes envolvidos na adesão / invasão.
Além disso, miRNA como miR-183/96/182 parece desempenhar um papel fundamental no ritmo circadiano .
Evolução
Os miRNAs são bem conservados em plantas e animais, e são considerados um componente vital e evolutivamente antigo da regulação gênica. Enquanto os componentes centrais da via do microRNA são conservados entre plantas e animais , repertórios de miRNA nos dois reinos parecem ter surgido independentemente com diferentes modos primários de ação.
microRNAs são marcadores filogenéticos úteis devido à sua taxa de evolução aparentemente baixa. Origem dos microRNAs como um mecanismo regulatório desenvolvido a partir de maquinários de RNAi anteriores que foram inicialmente usados como defesa contra material genético exógeno, como vírus. Sua origem pode ter permitido o desenvolvimento de inovações morfológicas e, ao tornar a expressão gênica mais específica e "sintonizável", permitiu a gênese de órgãos complexos e talvez, em última análise, vida complexa. Explosões rápidas de inovação morfológica são geralmente associadas a uma alta taxa de acúmulo de microRNA.
Novos microRNAs são criados de várias maneiras. Os novos microRNAs podem se originar da formação aleatória de grampos em seções "não codificantes" do DNA (ou seja, íntrons ou regiões intergênicas), mas também pela duplicação e modificação dos microRNAs existentes. Os microRNAs também podem se formar a partir de duplicações invertidas de sequências codificadoras de proteínas, o que permite a criação de uma estrutura em gancho dobrável. A taxa de evolução (isto é, substituição de nucleotídeos) em microRNAs recentemente originados é comparável àquela em outras partes do DNA não codificante, implicando em evolução por deriva neutra; no entanto, os microRNAs mais antigos têm uma taxa de mudança muito menor (geralmente menos de uma substituição a cada cem milhões de anos), sugerindo que, uma vez que um microRNA ganha uma função, ele passa por uma seleção purificadora. Regiões individuais dentro de um gene miRNA enfrentam diferentes pressões evolutivas, onde regiões que são vitais para processamento e função têm níveis mais altos de conservação. Neste ponto, um microRNA raramente é perdido do genoma de um animal, embora microRNAs mais novos (portanto, presumivelmente não funcionais) sejam freqüentemente perdidos. Em Arabidopsis thaliana , o fluxo líquido de genes miRNA foi previsto entre 1,2 e 3,3 genes por milhão de anos. Isso os torna um marcador filogenético valioso, e eles estão sendo vistos como uma possível solução para problemas filogenéticos pendentes, como o relacionamento dos artrópodes . Por outro lado, em vários casos, os microRNAs se correlacionam mal com a filogenia, e é possível que sua concordância filogenética reflita amplamente uma amostra limitada de microRNAs.
Os microRNAs estão presentes nos genomas da maioria dos organismos eucarióticos, desde as algas marrons até os animais. No entanto, a diferença em como esses microRNAs funcionam e a maneira como são processados sugere que os microRNAs surgiram de forma independente em plantas e animais.
Com foco nos animais, o genoma de Mnemiopsis leidyi parece não ter microRNAs reconhecíveis, bem como as proteínas nucleares Drosha e Pasha , que são críticas para a biogênese de microRNAs canônicos. É o único animal até agora relatado como desaparecido de Drosha. MicroRNAs desempenham um papel vital na regulação da expressão gênica em todos os animais não ctenóforos investigados até agora, exceto para Trichoplax adhaerens , o único membro conhecido do filo Placozoa .
Em todas as espécies, mais de 5.000 miRNAs diferentes foram identificados em março de 2010. Embora sequências curtas de RNA (50 - centenas de pares de bases) de uma função amplamente comparável ocorram em bactérias, as bactérias não possuem microRNAs verdadeiros.
Detecção e manipulação experimental
Enquanto os pesquisadores se concentraram na expressão de miRNA em processos fisiológicos e patológicos, várias variáveis técnicas relacionadas ao isolamento de microRNA emergiram. A estabilidade das amostras de miRNA armazenadas tem sido questionada. microRNAs degradam-se muito mais facilmente do que mRNAs, em parte devido ao seu comprimento, mas também por causa de RNases ubiquamente presentes . Isso torna necessário resfriar as amostras no gelo e usar equipamento sem RNase .
A expressão de microRNA pode ser quantificada em um processo de reação em cadeia da polimerase de duas etapas de RT-PCR modificado seguido por PCR quantitativo . As variações deste método alcançam a quantificação absoluta ou relativa. miRNAs também podem ser hibridizados para microarrays , slides ou chips com sondas para centenas ou milhares de alvos de miRNA, de modo que os níveis relativos de miRNAs podem ser determinados em diferentes amostras. Os microRNAs podem ser descobertos e perfilados por métodos de sequenciamento de alto rendimento ( sequenciamento de microRNA ). A atividade de um miRNA pode ser inibida experimentalmente usando um oligo de ácido nucleico bloqueado (LNA) , um oligo de Morfolino ou um oligo de RNA de 2'-O-metil. Um miRNA específico pode ser silenciado por um antagomir complementar . A maturação do microRNA pode ser inibida em vários pontos por oligos bloqueadores estereoquímicos. O sítio de miRNA alvo de um transcrito de mRNA também pode ser bloqueado por um oligo bloqueador estérico. Para a detecção "in situ" de miRNA, podem ser utilizadas sondas LNA ou Morpholino. A conformação bloqueada de LNA resulta em propriedades de hibridização aprimoradas e aumenta a sensibilidade e seletividade, tornando-o ideal para a detecção de miRNA curto.
A quantificação de alto rendimento de miRNAs é propensa a erros, para a maior variação (em comparação com mRNAs ) que vem com problemas metodológicos. A expressão de mRNA é, portanto, frequentemente analisada para verificar os efeitos de miRNA em seus níveis (por exemplo, em). Os bancos de dados podem ser usados para emparelhar mRNA - e miRNA-dados que predizem miRNA-alvos com base em sua sequência de base. Embora isso geralmente seja feito após a detecção de miRNAs de interesse (por exemplo, por causa dos altos níveis de expressão), foram propostas ideias para ferramentas de análise que integram mRNA - e informações de expressão de miRNA.
Doença
Assim como o miRNA está envolvido no funcionamento normal das células eucarióticas, a desregulação do miRNA também está associada à doença. Um banco de dados disponível publicamente e com curadoria manual, miR2Disease, documenta as relações conhecidas entre a desregulação de miRNA e as doenças humanas.
Doenças hereditárias
Uma mutação na região da semente do miR-96 causa perda auditiva progressiva hereditária.
Uma mutação na região da semente do miR-184 causa ceratocone hereditário com catarata polar anterior.
A exclusão do cluster miR-17 ~ 92 causa defeitos esqueléticos e de crescimento.
Câncer
A primeira doença humana conhecida por estar associada à desregulação de miRNA foi a leucemia linfocítica crônica. Muitos outros miRNAs também têm ligações com o câncer e, portanto, às vezes são chamados de " oncomirs ". Em células B malignas, miRNAs participam de vias fundamentais para o desenvolvimento de células B, como sinalização do receptor de células B (BCR), migração / adesão de células B, interações célula-célula em nichos imunológicos e a produção e troca de classe de imunoglobulinas. MiRNAs influenciam a maturação das células B, geração de células B pré, zona marginal, folicular, B1, plasma e memória.
Outro papel do miRNA em cânceres é usar seu nível de expressão para o prognóstico. Em amostras de NSCLC , níveis baixos de miR-324 a podem servir como um indicador de baixa sobrevivência. Tanto os níveis elevados de miR-185 quanto os baixos de miR-133b podem se correlacionar com metástases e baixa sobrevida no câncer colorretal .
Além disso, miRNAs específicos podem estar associados a certos subtipos histológicos de câncer colorretal. Por exemplo, os níveis de expressão de miR-205 e miR-373 mostraram estar aumentados em cânceres colorretais mucinosos e cânceres de cólon associados à colite ulcerativa produtores de mucina, mas não em adenocarcinoma esporádico do cólon que carece de componentes mucinosos. Estudos in vitro sugeriram que miR-205 e miR-373 podem induzir funcionalmente diferentes características de progressão neoplásica associada a mucinosas em células epiteliais intestinais.
A proliferação de células de carcinoma hepatocelular pode surgir da interação do miR-21 com MAP2K3, um gene repressor de tumor. O tratamento ideal para o câncer envolve a identificação precisa dos pacientes para terapia estratificada de risco. Aqueles com uma resposta rápida ao tratamento inicial podem se beneficiar de regimes de tratamento truncados, mostrando o valor de medidas precisas de resposta à doença. Os miRNAs circulantes livres de células (cimiRNAs) são altamente estáveis no sangue, são superexpressos no câncer e são quantificáveis no laboratório de diagnóstico. No linfoma de Hodgkin clássico , plasma miR-21, miR-494 e miR-1973 são biomarcadores de resposta à doença promissores. Os miRNAs circulantes têm o potencial de auxiliar na tomada de decisões clínicas e auxiliar na interpretação da tomografia por emissão de pósitrons combinada com a tomografia computadorizada . Eles podem ser realizados em cada consulta para avaliar a resposta da doença e detectar recidivas.
MicroRNAs têm potencial para serem usados como ferramentas ou alvos para o tratamento de diferentes tipos de câncer. O microRNA específico, miR-506 foi encontrado para funcionar como um antagonista de tumor em vários estudos. Foi descoberto que um número significativo de amostras de câncer cervical apresentou expressão negativa de miR-506. Além disso, miR-506 trabalha para promover a apoptose de células de câncer cervical, por meio de seu fator de transcrição da via hedgehog alvo direto, Gli3.
Reparo de DNA e câncer
O câncer é causado pelo acúmulo de mutações de danos ao DNA ou erros não corrigidos na replicação do DNA . Defeitos no reparo do DNA causam o acúmulo de mutações, que podem levar ao câncer. Vários genes envolvidos no reparo do DNA são regulados por microRNAs.
Mutações na linha germinativa em genes de reparo de DNA causam apenas 2–5% dos casos de câncer de cólon . No entanto, a expressão alterada de microRNAs, causando deficiências no reparo do DNA, está frequentemente associada a cânceres e pode ser um importante fator causal . Entre 68 cânceres de cólon esporádicos com expressão reduzida da proteína de reparo de incompatibilidade de DNA MLH1 , a maioria foi considerada deficiente devido à metilação epigenética da ilha CpG do gene MLH1 . No entanto, até 15% das deficiências de MLH1 em cânceres de cólon esporádicos pareceram ser devido à superexpressão do microRNA miR-155, que reprime a expressão de MLH1.
Em 29–66% dos glioblastomas , o reparo do DNA é deficiente devido à metilação epigenética do gene MGMT , que reduz a expressão proteica do MGMT. No entanto, para 28% dos glioblastomas, a proteína MGMT é deficiente, mas o promotor MGMT não é metilado. Em glioblastomas sem promotores MGMT metilados, o nível de microRNA miR-181d está inversamente correlacionado com a expressão da proteína de MGMT e o alvo direto de miR-181d é o MGMT mRNA 3'UTR (a região não traduzida de três primos do MGMT mRNA). Assim, em 28% dos glioblastomas, o aumento da expressão de miR-181d e a redução da expressão da enzima de reparo de DNA MGMT pode ser um fator causal.
As proteínas HMGA (HMGA1a, HMGA1b e HMGA2) estão implicadas no câncer e a expressão dessas proteínas é regulada por microRNAs. A expressão de HMGA é quase indetectável em tecidos adultos diferenciados, mas é elevada em muitos cânceres. As proteínas HMGA são polipeptídeos de ~ 100 resíduos de aminoácidos caracterizados por uma organização de sequência modular. Essas proteínas têm três regiões altamente carregadas positivamente, chamadas de ganchos AT , que se ligam ao sulco menor de trechos de DNA ricos em AT em regiões específicas do DNA. Neoplasias humanas, incluindo carcinomas da tireóide, próstata, cervical, colorretal, pancreático e ovariano, mostram um forte aumento das proteínas HMGA1a e HMGA1b. Camundongos transgênicos com HMGA1 direcionado a células linfóides desenvolvem linfoma agressivo, mostrando que a alta expressão de HMGA1 está associada a cânceres e que HMGA1 pode atuar como um oncogene. A proteína HMGA2 tem como alvo específico o promotor de ERCC1 , reduzindo assim a expressão deste gene de reparo de DNA. A expressão da proteína ERCC1 foi deficiente em 100% dos 47 cânceres de cólon avaliados (embora a extensão em que HGMA2 estava envolvida não seja conhecida).
Polimorfismos de nucleotídeo único (SNPs) podem alterar a ligação de miRNAs em 3'UTRs, por exemplo, o caso de hsa-mir181a e hsa-mir181b no gene supressor de tumor CDON.
Doença cardíaca
O papel global da função do miRNA no coração foi abordado pela inibição condicional da maturação do miRNA no coração murino . Isso revelou que os miRNAs desempenham um papel essencial durante o seu desenvolvimento. Estudos de perfil de expressão de miRNAs demonstram que os níveis de expressão de miRNAs específicos mudam em corações humanos doentes, apontando para seu envolvimento em cardiomiopatias . Além disso, estudos em animais sobre miRNAs específicos identificaram papéis distintos para miRNAs durante o desenvolvimento do coração e em condições patológicas, incluindo a regulação de fatores-chave importantes para a cardiogênese, a resposta de crescimento hipertrófico e a condutância cardíaca. Outro papel do miRNA em doenças cardiovasculares é usar seus níveis de expressão para diagnóstico, prognóstico ou estratificação de risco. Os miRNAs em modelos animais também foram associados ao metabolismo e à regulação do colesterol.
miRNA-712
O microRNA-712 murino é um biomarcador potencial (ou seja, preditor) para aterosclerose , uma doença cardiovascular da parede arterial associada à retenção de lipídios e inflamação. O fluxo sanguíneo não laminar também se correlaciona com o desenvolvimento de aterosclerose, pois os mecanossensores das células endoteliais respondem à força de cisalhamento do fluxo perturbado (fluxo d). Vários genes pró-aterogênicos, incluindo metaloproteinases de matriz (MMPs), são regulados positivamente pelo d-flow, mediando sinais pró-inflamatórios e pró-angiogênicos. Esses achados foram observados em artérias carótidas ligadas de camundongos para imitar os efeitos do fluxo-d. Dentro de 24 horas, miR-712 imaturo pré-existente formou miR-712 maduro, sugerindo que miR-712 é sensível ao fluxo. Coincidindo com esses resultados, o miR-712 também é regulado positivamente nas células endoteliais expostas ao fluxo-d que ocorre naturalmente na grande curvatura do arco aórtico.
Origem
A sequência pré-mRNA de miR-712 é gerada a partir do gene RN45s ribossomal murino na região espaçadora transcrita interna 2 (ITS2). XRN1 é uma exonuclease que degrada a região ITS2 durante o processamento de RN45s. A redução de XRN1 sob condições de fluxo d , portanto, leva ao acúmulo de miR-712.
Mecanismo
MiR-712 tem como alvo o inibidor de tecido de metaloproteinases 3 (TIMP3). Os TIMPs normalmente regulam a atividade das metaloproteinases da matriz (MMPs) que degradam a matriz extracelular (ECM). A ECM arterial é composta principalmente por fibras de colágeno e elastina , fornecendo o suporte estrutural e as propriedades de recuo das artérias. Essas fibras desempenham um papel crítico na regulação da inflamação e permeabilidade vascular, que são importantes no desenvolvimento da aterosclerose. Expresso pelas células endoteliais, o TIMP3 é o único TIMP ligado ao ECM. Uma diminuição na expressão de TIMP3 resulta em um aumento da degradação de ECM na presença de d-flow. Consistente com esses achados, a inibição do pré-miR712 aumenta a expressão de TIMP3 nas células, mesmo quando exposto a fluxo turbulento.
O TIMP3 também diminui a expressão de TNFα (um regulador pró-inflamatório) durante o fluxo turbulento. A atividade do TNFα em fluxo turbulento foi medida pela expressão da enzima conversora do TNFα (TACE) no sangue. TNFα diminuiu se miR-712 foi inibido ou TIMP3 superexpresso, sugerindo que miR-712 e TIMP3 regulam a atividade de TACE em condições de fluxo turbulento.
O anti-miR-712 suprime efetivamente a expressão de miR-712 induzida por d-flow e aumenta a expressão de TIMP3. O anti-miR-712 também inibe a hiperpermeabilidade vascular, reduzindo significativamente o desenvolvimento de lesões de aterosclerose e a infiltração de células imunes.
MicroRNA-205 homólogo humano
O homólogo humano de miR-712 foi encontrado no gene homólogo RN45s, que mantém miRNAs semelhantes aos de camundongos. MiR-205 de humanos compartilham sequências semelhantes com miR-712 de camundongos e é conservado na maioria dos vertebrados. MiR-205 e miR-712 também compartilham mais de 50% dos alvos de sinalização celular, incluindo TIMP3.
Quando testado, o d-flow diminuiu a expressão de XRN1 em humanos como fez em células endoteliais de camundongos, indicando um papel potencialmente comum de XRN1 em humanos.
Doenca renal
A deleção direcionada de Dicer nas células progenitoras renais derivadas de FoxD1 em um modelo murino resultou em um fenótipo renal complexo, incluindo expansão de progenitores de néfron , menos células de renina , arteríolas de músculo liso , perda mesangial progressiva e aneurismas glomerulares. Perfil de transcriptoma completo de alto rendimento do modelo de camundongo nocaute FoxD1-Dicer revelou suprarregulação ectópica do gene pró-apoptótico, Bcl2L11 (Bim) e desregulação da via p53 com aumento nos genes efetores de p53, incluindo Bax , Trp53inp1 , Jun, Cdkn1a , Mmp2 e Arid3a . Os níveis de proteína p53 permaneceram inalterados, sugerindo que os miRNAs do estroma FoxD1 reprimem diretamente os genes efetores de p53. Usando uma abordagem de rastreamento de linhagem seguida por classificação de células ativadas por fluorescência , o perfil de miRNA das células derivadas de FoxD1 não apenas definiu de forma abrangente a paisagem transcricional de miRNAs que são críticos para o desenvolvimento vascular, mas também identificou miRNAs chave que são susceptíveis de modular o fenótipo renal em sua ausência. Esses miRNAs incluem miRs ‐ 10a, 18a, 19b, 24, 30c, 92a, 106a, 130a, 152, 181a, 214, 222, 302a, 370 e 381 que regulam Bcl2L11 (Bim) e miRs ‐ 15b, 18a, 21, 30c, 92a, 106a, 125b ‐ 5p, 145, 214, 222, 296‐5p e 302a que regulam os genes efetores de p53. Consistente com os resultados do perfil, a apoptose ectópica foi observada nos derivados celulares da linhagem progenitora derivada de FoxD1 e reitera a importância dos miRNAs do estroma renal na homeostase celular.
Sistema nervoso
Os miRNAs parecem regular o desenvolvimento e a função do sistema nervoso . MiARNs neurais estão envolvidos em vários estágios de desenvolvimento, incluindo sináptica dendritogenesis (envolvendo o miR-132 , miR-134 e miR-124 ), sinapse formação e maturação de sinapses (em que o miR-134 e miR-138 são pensados para ser envolvido). A eliminação da formação de miRNA em camundongos pelo silenciamento experimental de Dicer levou a resultados patológicos, como redução do tamanho neuronal e anormalidades motoras quando silenciados em neurônios estriados e neurodegeneração quando silenciados em neurônios do prosencéfalo . Alguns estudos encontram expressão alterada de miRNA na doença de Alzheimer , bem como esquizofrenia , transtorno bipolar , depressão maior e transtornos de ansiedade .
Golpe
De acordo com o Centro de Controle e Prevenção de Doenças, o AVC é uma das principais causas de morte e invalidez de longo prazo na América. 87% dos casos são acidentes vasculares cerebrais isquêmicos, que resultam de um bloqueio na artéria do cérebro que transporta sangue rico em oxigênio. A obstrução do fluxo sanguíneo significa que o cérebro não pode receber os nutrientes necessários, como oxigênio e glicose, e remover resíduos, como dióxido de carbono. Os miRNAs desempenham um papel no silenciamento de genes pós-tradução ao direcionar genes na patogênese da isquemia cerebral, como a via inflamatória, angiogênese e apoptótica.
Alcoolismo
O papel vital dos miRNAs na expressão gênica é significativo para o vício , especificamente o alcoolismo . O abuso crônico de álcool resulta em mudanças persistentes na função cerebral mediadas em parte por alterações na expressão gênica . A regulação global por miRNA de muitos genes a jusante considera significativa em relação à reorganização ou conexões sinápticas ou adaptações neurais de longo prazo envolvendo a mudança comportamental do consumo de álcool para a abstinência e / ou dependência. Descobriu-se que até 35 diferentes miRNAs estão alterados no cérebro pós-morte alcoólico, todos os quais têm como alvo genes que incluem a regulação do ciclo celular , apoptose , adesão celular , desenvolvimento do sistema nervoso e sinalização celular . Níveis alterados de miRNA foram encontrados no córtex pré-frontal medial de camundongos dependentes de álcool, sugerindo o papel do miRNA na orquestração de desequilíbrios traducionais e na criação de proteínas expressas diferencialmente dentro de uma área do cérebro onde o comportamento cognitivo complexo e a tomada de decisão provavelmente se originam.
Os miRNAs podem ser regulados positivamente ou regulados negativamente em resposta ao uso crônico de álcool. A expressão do miR-206 aumentou no córtex pré-frontal de ratos dependentes de álcool, visando o fator de transcrição do fator neurotrófico derivado do cérebro ( BDNF ) e, finalmente, reduzindo sua expressão. O BDNF desempenha um papel crítico na formação e maturação de novos neurônios e sinapses, sugerindo uma possível implicação no crescimento de sinapses / plasticidade sináptica em usuários de álcool. O miR-155, importante na regulação das respostas de neuroinflamação induzida pelo álcool , foi encontrado para ser regulado positivamente, sugerindo o papel da microglia e das citocinas inflamatórias na fisiopatologia do álcool. A regulação negativa do miR-382 foi encontrada no núcleo accumbens , uma estrutura no prosencéfalo basal significativa na regulação dos sentimentos de recompensa que alimentam os hábitos motivacionais. miR-382 é o alvo para o receptor de dopamina D1 (DRD1), e sua superexpressão resulta na suprarregulação de DRD1 e delta fosB , um fator de transcrição que ativa uma série de eventos de transcrição no núcleo accumbens que, em última análise, resultam em comportamentos aditivos. Alternativamente, a superexpressão de miR-382 resultou em beber atenuado e na inibição da suprarregulação de DRD1 e delta fosB em modelos de alcoolismo em ratos, demonstrando a possibilidade de usar fármacos direcionados a miRNA em tratamentos.
Obesidade
miRNAs desempenham papéis cruciais na regulação da diferenciação de progenitores de células-tronco em adipócitos . Estudos para determinar qual papel as células-tronco pluripotentes desempenham na adipogênese , foram examinados na linha de células estromais derivadas da medula óssea humana imortalizada hMSC-Tert20. A expressão diminuída de miR-155 , miR-221 e miR-222 foi encontrada durante a programação adipogênica de hMSCs imortalizadas e primárias, sugerindo que eles agem como reguladores negativos da diferenciação. Por outro lado, a expressão ectópica dos miRNAs 155 , 221 e 222 inibiu significativamente a adipogênese e reprimiu a indução dos reguladores principais PPARγ e CCAAT / proteína alfa de ligação ao intensificador ( CEBPA ). Isso abre caminho para possíveis tratamentos genéticos para a obesidade.
Outra classe de miRNAs que regulam a resistência à insulina , obesidade e diabetes é a família let-7 . O Let-7 se acumula nos tecidos humanos durante o envelhecimento . Quando let-7 foi superexpresso ectopicamente para imitar o envelhecimento acelerado, os camundongos se tornaram resistentes à insulina e, portanto, mais propensos a obesidade induzida por dieta rica em gordura e diabetes . Em contraste, quando let-7 foi inibido por injeções de antagomirs específicos para let-7 , os camundongos se tornaram mais sensíveis à insulina e notavelmente resistentes à obesidade induzida por dieta rica em gordura e diabetes. A inibição do let-7 não só pode prevenir a obesidade e o diabetes, mas também pode reverter e curar a doença. Essas descobertas experimentais sugerem que a inibição do let-7 pode representar uma nova terapia para obesidade e diabetes tipo 2.
Hemostasia
Os miRNAs também desempenham papéis cruciais na regulação de cascatas enzimáticas complexas, incluindo o sistema hemostático de coagulação do sangue. Estudos em grande escala de direcionamento de miRNA funcional descobriram recentemente alvos terapêuticos lógicos no sistema hemostático.
RNAs não codificantes
Quando o projeto do genoma humano mapeou seu primeiro cromossomo em 1999, previu-se que o genoma conteria mais de 100.000 genes codificadores de proteínas. No entanto, apenas cerca de 20.000 foram identificados. Desde então, o advento de abordagens de bioinformática combinadas com estudos de genoma examinando o transcriptoma, sequenciamento sistemático de bibliotecas de cDNA de comprimento total e validação experimental (incluindo a criação de oligonucleotídeos antisense derivados de miRNA chamados antagomirs ) revelaram que muitos transcritos não são codificadores de proteínas RNA, incluindo vários snoRNAs e miRNAs.
Vírus
Os microRNAs virais desempenham um papel importante na regulação da expressão gênica de genes virais e / ou hospedeiros para beneficiar o vírus. Portanto, miRNAs desempenham um papel fundamental nas interações vírus-hospedeiro e na patogênese de doenças virais. Acredita-se que a expressão de ativadores de transcrição pelo DNA do herpesvírus-6 humano seja regulada por miRNA viral.
Previsão de alvo
miRNAs podem se ligar a transcritos de RNA mensageiro alvo (mRNA) de genes codificadores de proteínas e controlar negativamente sua tradução ou causar degradação de mRNA. É de fundamental importância identificar os alvos de miRNA com precisão. Uma comparação do desempenho preditivo de dezoito algoritmos in silico está disponível. Estudos em grande escala de direcionamento de miRNA funcional sugerem que muitos miRNAs funcionais podem ser perdidos por algoritmos de predição de alvo.
Veja também
- Oligonucleotídeos anti-miRNA
- Expressão genetica
- Lista de ferramentas de predição de genes de miRNA
- Lista de ferramentas de previsão de alvo miRNA
- MicroDNA
- miR-324-5p
- Interferência de RNA
- RNA de interferência pequeno
- MicroRNA derivado de RNA nucleolar pequeno
Referências
Leitura adicional
- Definição e classificação de miRNA: Ambros V, Bartel B, Bartel DP, Burge CB, Carrington JC, Chen X, Dreyfuss G, Eddy SR, Griffiths-Jones S, Marshall M, Matzke M, Ruvkun G, Tuschl T (março de 2003). "Um sistema uniforme para anotação de microRNA" . RNA . 9 (3): 277–9. doi : 10.1261 / rna.2183803 . PMC 1370393 . PMID 12592000 .
- Revisão científica de RNA pequeno: Baulcombe D (setembro de 2002). "Eventos de DNA. Um microcosmo de RNA". Ciência . 297 (5589): 2002–3. doi : 10.1126 / science.1077906 . PMID 12242426 . S2CID 82531727 .
- Descoberta de lin-4 , o primeiro miRNA a ser descoberto: Lee RC, Feinbaum RL, Ambros V (dezembro de 1993). "O gene heterocrônico lin-4 de C. elegans codifica pequenos RNAs com complementaridade antisense ao lin-14" . Cell . 75 (5): 843–54. doi : 10.1016 / 0092-8674 (93) 90529-Y . PMID 8252621 .
links externos
- O banco de dados miRBase
- miRTarBase , o banco de dados de interações microRNA-alvo validado experimentalmente.
- semirna , aplicativo da Web para pesquisar microRNAs em um genoma de planta.
- ONCO.IO : Recurso integrativo para análise de microRNA e fatores de transcrição no câncer.
- MirOB : MicroRNA visa banco de dados e análise de dados e ferramenta de dataviz.
- Banco de dados ChIPBase : um banco de dados de acesso aberto para decodificar os fatores de transcrição que estiveram envolvidos ou afetaram a transcrição de microRNAs de dados ChIP-seq.
- Um vídeo animado do processo de biogênese do microRNA .
- Reagentes de modulação de miRNA para permitir a regulação positiva ou supressão da função de microRNA maduro endógeno